cellchat-(1)单数据集的细胞通讯分析
原创学习,记录,分享。
上一篇讲了cellchat安装和数据准备,接下来进行单数据集的细胞通讯分析。
1 创建cellchat对象
这里直接通过上次保存的Seurat数据生成。
library(tidyverse)
library(Seurat)
library(CellChat)
pbmc3k?<-?read_rds('data/pbmc3k_anno.rds')
#?加个label
pbmc3k$label_cc?<-?Idents(pbmc3k)
#?创建cellchat对象,注意需要用Normalized?data,?这里用cellchat提供的`normalizeData`函数
cellchat?<-?createCellChat(normalizeData(pbmc3k[['RNA']]@counts),?
???????????????????????????meta?=?pbmc3k[[]],
???????????????????????????group.by?=?'label_cc')[1] "Create a CellChat object from a data matrix" Set cell identities for the new CellChat object The cell groups used for CellChat analysis are Naive CD4 T CD14+ Mono Memory CD4 T B CD8 T FCGR3A+ Mono NK DC Platelet
可以看到已经创建成功,并且打印出了cellchat对象信息。
2 选择数据库
接下来需要选择配体受体数据库,cellchat的数据库是基于文献手动生成的,包含的物种和数量为:
human1939个mouse2021个zebrafish2774个
相对来说还是有些少的,不过也可以根据需要自己增减,后续会讲到如何更新数据库。 数据库中的配体受体对根据相互作用的类型分为:
分泌型如各种细胞因子的生长趋化作用;细胞外基质受体如整联蛋白介导的细胞与细胞外基质的黏着;细胞间接触型如钙黏蛋白和选择素等介导的细胞和细胞间的粘附。
另外还根据聚合形式和这些受体配体的信息来源进行了分类,可以用下面代码查看:
showDatabaseCategory(CellChatDB.human)?#替换后缀的物种名即可查看其他物种的分类情况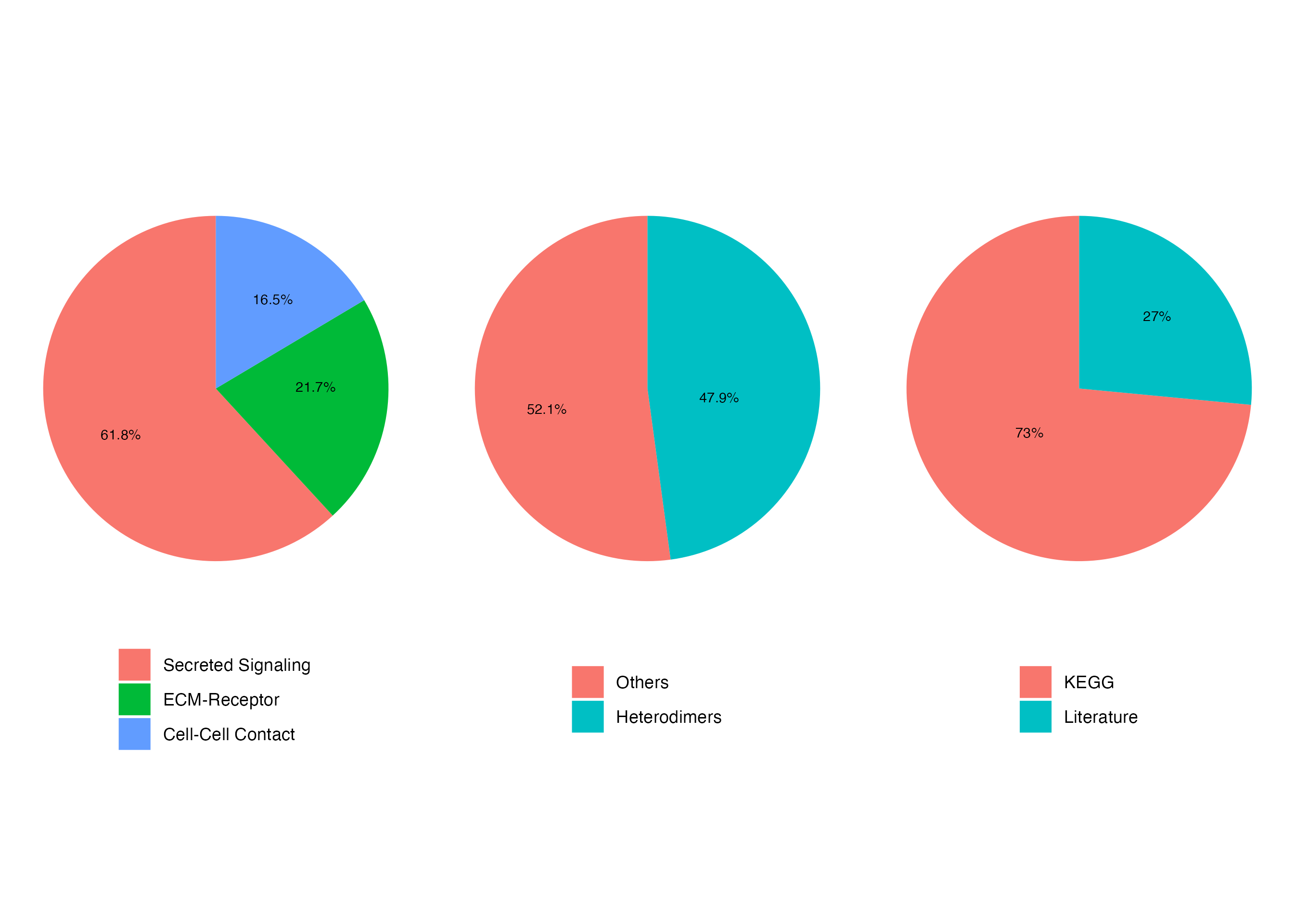
也可用以下代码查看其中的相互作用信息
glimpse(CellChatDB.human$interaction)
#以下是终端输出内容,为了方便浏览放到了代码框中
Rows:?1,939
Columns:?11
$?interaction_name???<chr>?"TGFB1_TGFBR1_TGFBR2",?"TGFB2_TGFBR1_TGFBR2",?"TGFB3_TGFBR1_TGFBR2",?"…
$?pathway_name???????<chr>?"TGFb",?"TGFb",?"TGFb",?"TGFb",?"TGFb",?"TGFb",?"TGFb",?"TGFb",?"TGFb"…
$?ligand?????????????<chr>?"TGFB1",?"TGFB2",?"TGFB3",?"TGFB1",?"TGFB1",?"TGFB2",?"TGFB2",?"TGFB3"…
$?receptor???????????<chr>?"TGFbR1_R2",?"TGFbR1_R2",?"TGFbR1_R2",?"ACVR1B_TGFbR2",?"ACVR1C_TGFbR2…
$?agonist????????????<chr>?"TGFb?agonist",?"TGFb?agonist",?"TGFb?agonist",?"TGFb?agonist",?"TGFb?…
$?antagonist?????????<chr>?"TGFb?antagonist",?"TGFb?antagonist",?"TGFb?antagonist",?"TGFb?antagon…
$?co_A_receptor??????<chr>?"",?"",?"",?"",?"",?"",?"",?"",?"",?"",?"",?"",?"",?"",?"",?"",?"",?""…
$?co_I_receptor??????<chr>?"TGFb?inhibition?receptor",?"TGFb?inhibition?receptor",?"TGFb?inhibiti…
$?evidence???????????<chr>?"KEGG:?hsa04350",?"KEGG:?hsa04350",?"KEGG:?hsa04350",?"PMID:?27449815"…
$?annotation?????????<chr>?"Secreted?Signaling",?"Secreted?Signaling",?"Secreted?Signaling",?"Sec…
$?interaction_name_2?<chr>?"TGFB1?-?(TGFBR1+TGFBR2)",?"TGFB2?-?(TGFBR1+TGFBR2)",?"TGFB3?-?(TGFBR1…接着分析步骤,先在cellchat对象中添加需要的数据库,这里因为pbmc3k是人的细胞,选择人的数据库
cellchat@DB?<-?CellChatDB.human
#也可以只选择其中某种类型的相互作用,如:
#?cellchat@DB?<-?subsetDB(CellChatDB.human,?search?=?'Secreted?Signaling')3 预处理表达数据
细胞间是否有通讯作用是基于配体或者受体是否过表达来推断的,所以需先进行过表达基因的鉴定:
#?首先取一下要计算的基因子集,因为CellChatDB.human中包含4万多基因,
#?而我们的pbmc3k中只有1万多基因,这样可以节省接下来的计算时间
cellchat?<-?subsetData(cellchat)
future::plan("multisession",?workers?=?4)?#设置使用的电脑核心数,并行计算节省时间
#?注意,windows系统需将'multisession'改为'multiprocess'
cellchat?<-?identifyOverExpressedGenes(cellchat)
cellchat?<-?identifyOverExpressedInteractions(cellchat)4 推断相互作用网络
预处理完表达值后,cellchat会计算相互作用的可能性和相互作用网络:
#?trim参数用来过滤掉只在少量细胞表达的基因,
#?0.1表示如果某基因在一种细胞类型中表达比例少于10%,
#?则该基因在这种细胞中平均表达量归0,也就是该配体/受体不在这种细胞中表达
#?population.size设置为TRUE表示需要考虑每种细胞类型中细胞数量的多少,对于分选的细胞需要设置为FALSE
cellchat?<-?computeCommunProb(cellchat,?trim?=?0.1,?population.size?=?T)?
#?min.cells用来过滤掉细胞数目较少的细胞类型
cellchat?<-?filterCommunication(cellchat,?min.cells?=?10)
#?在信号通路水平推断相互作用
cellchat?<-?computeCommunProbPathway(cellchat)
cellchat?<-?aggregateNet(cellchat)到这里细胞通讯的推断已经完成,所有数据都存储在了cellchat对象中,接下来就是可视化了。
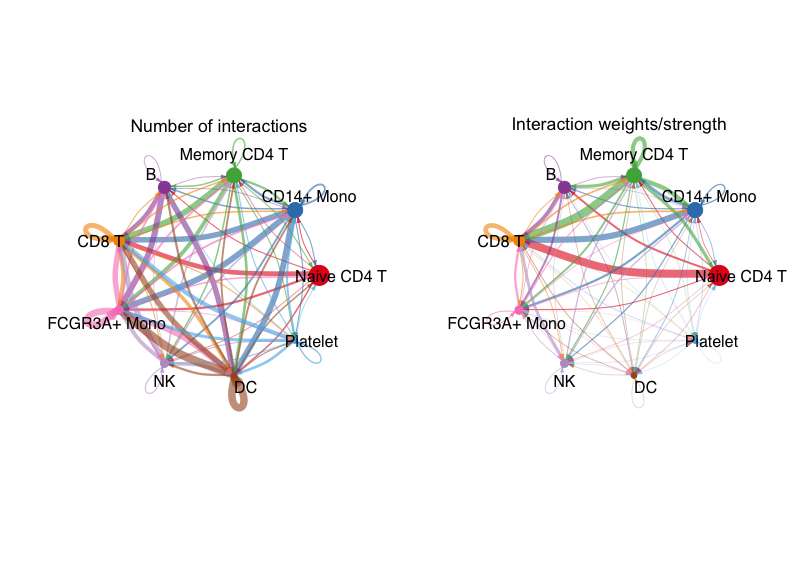
总的相互作用网络:
groupSize?<-?as.numeric(table(cellchat@idents))
par(mfrow?=?c(1,2),?xpd=TRUE)
netVisual_circle(
??cellchat@net$count,
??vertex.weight?=?groupSize,
??weight.scale?=?T,
??label.edge?=?F,
??title.name?=?"Number?of?interactions"
)
netVisual_circle(
??cellchat@net$weight,
??vertex.weight?=?groupSize,
??weight.scale?=?T,
??label.edge?=?F,
??title.name?=?"Interaction?weights/strength"
)
每种细胞分别作为source的相互作用网络:
mat?<-?cellchat@net$weight
par(mfrow?=?c(3,4),?xpd=TRUE)
for?(i?in?1:nrow(mat))?{
??mat2?<-?matrix(0,?nrow?=?nrow(mat),?ncol?=?ncol(mat),?dimnames?=?dimnames(mat))
??mat2[i,?]?<-?mat[i,?]
??netVisual_circle(mat2,?vertex.weight?=?groupSize,?weight.scale?=?T,?edge.weight.max?=?max(mat),?title.name?=?rownames(mat)[i])
}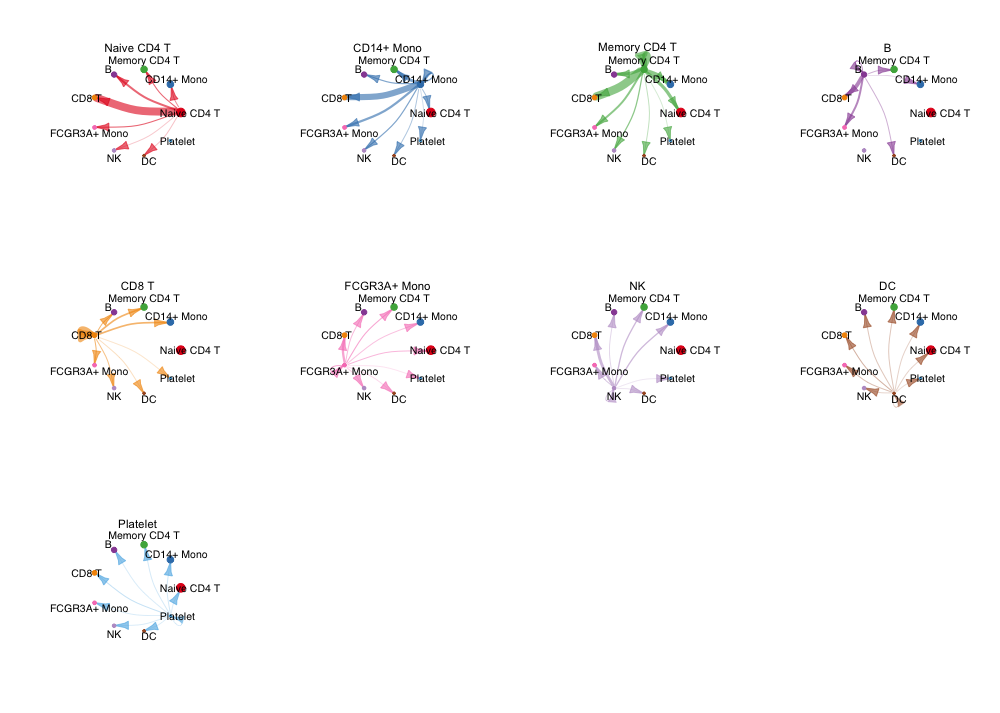
以上两种图只能看到一个大概的相互作用关系,满足不了科研故事的需求,后面会介绍不同的可视化方式以根据需要进行信息挖掘和数据展示。
参考资料:
- cellchat官方教程(https://github.com/sqjin/CellChat)
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
