scRNA分析| Seurat堆叠小提琴图不满足? 那就ggplot2 堆叠 各种元素
scRNA分析| Seurat堆叠小提琴图不满足? 那就ggplot2 堆叠 各种元素

单细胞常见的可视化方式有DimPlot,FeaturePlot ,DotPlot ,VlnPlot 和 DoHeatmap几种 ,Seurat均可以实现,但文献中的图大多会精美很多。比如
惊艳umap图: scRNA复现|所见即所得,和Cell学umap,plot1cell完成惊艳的细胞注释umap图;
DimPlot美化 scRNA分析 | 定制 美化FeaturePlot 图,你需要的都在这,
DotPlot美化scRNA分析| 和SCI学 定制化聚类点图(Dotplot ),含二行代码出图方式,
DoHeatmap 热图:scRNA分析| DoHeatmap 美化,dittoSeq ,scillus 一行代码出图,你PICK谁?
本次介绍Seurat 以及 ggplot2绘制,优化堆叠小提琴图的方法。
一 载入R包,数据
仍然使用之前注释过的sce.anno.RData数据 ,后台回复 anno 即可获取。
library(Seurat)
library(tidyverse)
load("sce.anno.RData")
head(sce2,2)
二 Seurat 调整,美化
1,基础VlnPlot图
首先计算marker基因,然后使用seurat的DoHeatmap 函数绘制初始热图
all_markers <- FindAllMarkers(object = sce2)
top5 <- all_markers %>%
group_by(cluster) %>%
top_n(5, avg_log2FC)
###少量基因
VlnPlot(sce2, features = c("CD3D","SPP1"))
### 所有marker 基因
VlnPlot(sce2, features = top5$gene)当展示少量基因时候,很清晰 。但是更常见的时候需要同时展示各个cluster/celltype的marker gene ,这时候就会看不清晰。

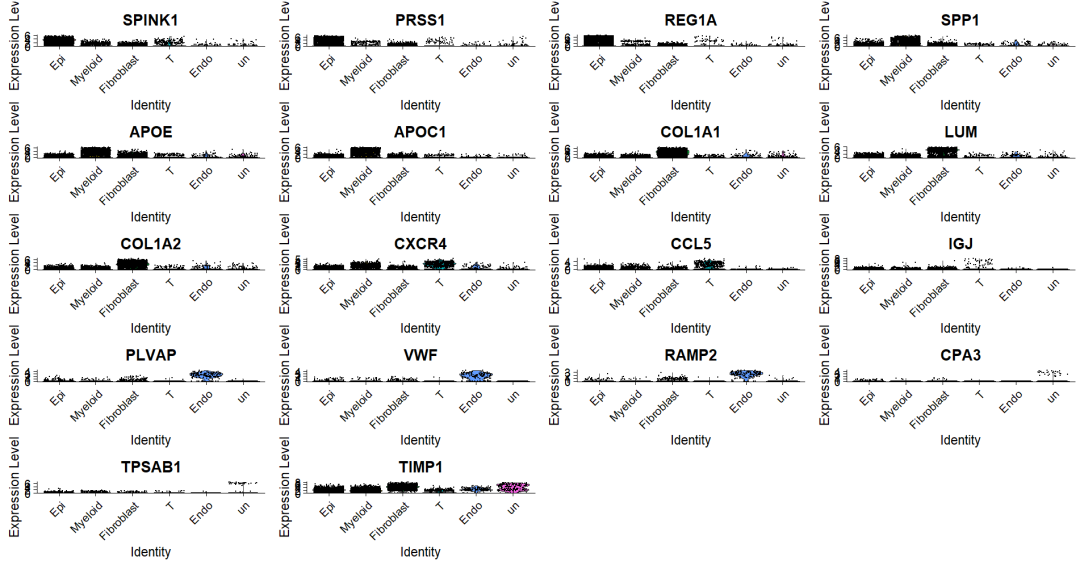
2,Seurat-堆叠VlnPlot图
Seurat的VlnPlot函数中stack 参数可以实现堆叠小提琴图,flip 是否翻转
#Seurat 的stack 函数
a <- VlnPlot(sce2, features = top5$gene, stack = TRUE, sort = TRUE) +
theme(legend.position = "none") + ggtitle("Identity on y-axis")
# flip 翻转
b <- VlnPlot(sce2, features = top5$gene, stack = TRUE, sort = TRUE, flip = TRUE) +
theme(legend.position = "none") + ggtitle("Identity on x-axis")
a + b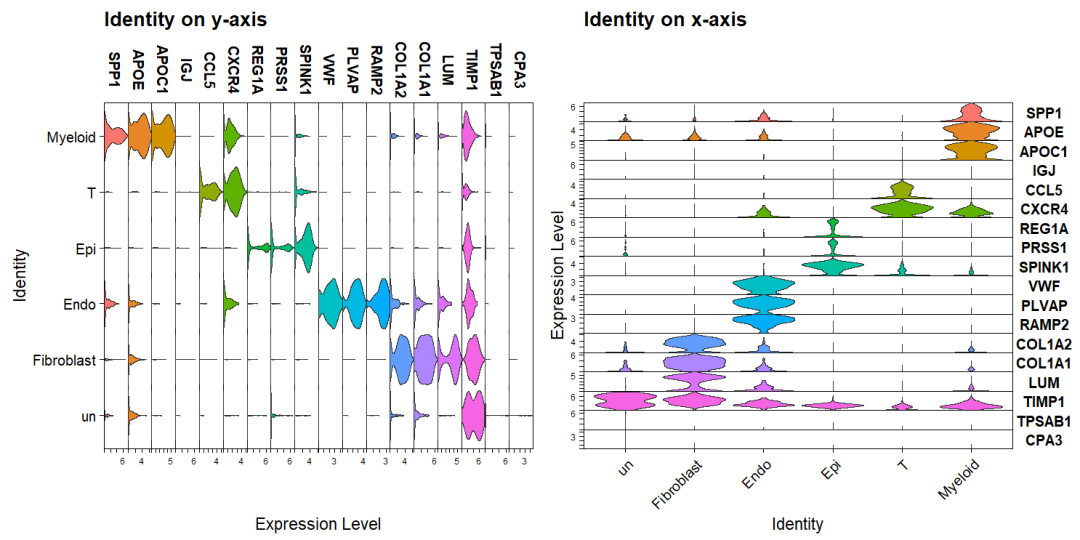
3,Seurat-优化颜色,大小,方向
自定义颜色,是否排序,主题等信息更是和前面的一样,直接添加theme信息即可。
注意如果想要每种cluster/celltype是一种颜色的话使用split.by参数。
my36colors <-c('#E5D2DD', '#53A85F', '#F1BB72', '#F3B1A0', '#D6E7A3', '#57C3F3', '#476D87',
'#E95C59', '#E59CC4', '#AB3282', '#23452F', '#BD956A', '#8C549C', '#585658',
'#9FA3A8', '#E0D4CA', '#5F3D69', '#C5DEBA', '#58A4C3', '#E4C755', '#F7F398',
'#AA9A59', '#E63863', '#E39A35', '#C1E6F3', '#6778AE', '#91D0BE', '#B53E2B',
'#712820', '#DCC1DD', '#CCE0F5', '#CCC9E6', '#625D9E', '#68A180', '#3A6963',
'#968175'
)
VlnPlot(sce2, features = top_marker$gene,
stack = TRUE,
sort = TRUE,
cols = my36colors,
split.by = "celltype" , #每种cluster 一个颜色
flip = TRUE) +
theme(legend.position = "none") +
ggtitle("Identity on x-axis")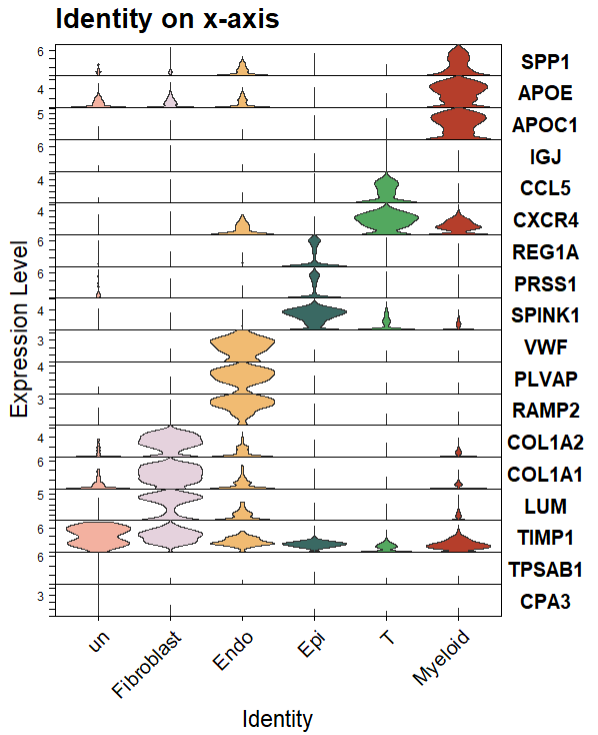
Seurat的堆叠小提琴图其实已经可以了,当然也可以使用ggplot2进行更多的自定义。
三 ggplot2-堆叠小提琴图
1,提取,转化数据
首先使用FetchData提取出marker gene的表达量,celltype /seurat_clusters(宽数据),然后转为ggplot2读取的长数据类型 。
此外对照上述的图,可以看到celltype /seurat_clusters一个表达量值,而FetchData得到的是每个cell 的表达量,因此还需要计算每种cluster的基因均值。
vln.dat=FetchData(sce2,c(top_marker$gene,"celltype","seurat_clusters"))
vln.dat$Cell <- rownames(vln.dat)
#宽转长
vln.dat.melt <- reshape2::melt(vln.dat, id.vars = c("Cell","seurat_clusters"),
measure.vars = top_marker$gene,
variable.name = "gene",
value.name = "Expr") %>%
group_by(seurat_clusters,gene) %>% #分组
mutate(fillcolor=mean(Expr)) #计算均值
2,ggplot2 绘制-核心
ggplot(vln.dat.melt, aes(factor(seurat_clusters), Expr, fill = gene)) +
geom_violin(scale = "width", adjust = 1, trim = TRUE) +
facet_grid(rows = vars(gene), scales = "free", switch = "y")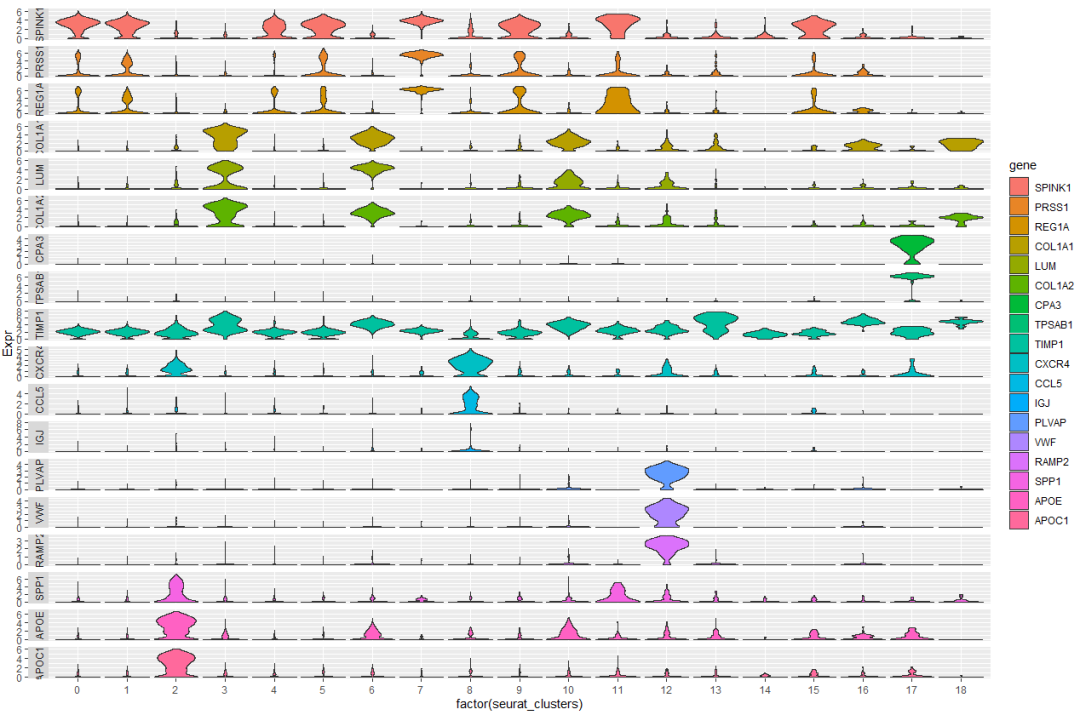
3,ggplot2 绘制-优化
上述是ggplot2绘制堆叠小提琴图的核心代码,可以做很多调整
(1)主题(大小,颜色),legend 等
(2)“翻转”(使用aes调整横纵坐标)
p1 <- ggplot(vln.dat.melt, aes(gene, Expr, fill = gene)) +
geom_violin(scale = "width", adjust = 1, trim = TRUE) +
scale_y_continuous(expand = c(0, 0), position="right", labels = function(x)
c(rep(x = "", times = length(x)-2), x[length(x) - 1], "")) +
facet_grid(rows = vars(seurat_clusters), scales = "free", switch = "y") +
scale_fill_manual(values = my36colors) +
theme_cowplot(font_size = 12) +
theme(legend.position = "none", panel.spacing = unit(0, "lines"),
plot.title = element_text(hjust = 0.5),
panel.background = element_rect(fill = NA, color = "black"),
plot.margin = margin(7, 7, 0, 7, "pt"),
strip.background = element_blank(),
strip.text = element_text(face = "bold"),
strip.text.y.left = element_text(angle = 0),
axis.title.x = element_blank(),
axis.ticks.x = element_blank(),
axis.text.x = element_text(angle = 90, hjust = 1, vjust = 0.5, color = "black")
) +
ggtitle("Feature on x-axis with annotation") + ylab("Expression Level")
p1
(3)添加基因的分组/注释
A:添加分组,注释
假设知道marker gene的通路,也可以添加上(为了美观先隐藏p1中的横坐标基因标签)
#隐藏axis.text.x
p2 <- ggplot(vln.dat.melt, aes(gene, Expr, fill = gene)) +
geom_violin(scale = "width", adjust = 1, trim = TRUE) +
scale_y_continuous(expand = c(0, 0), position="right", labels = function(x)
c(rep(x = "", times = length(x)-2), x[length(x) - 1], "")) +
facet_grid(rows = vars(seurat_clusters), scales = "free", switch = "y") +
scale_fill_manual(values = my36colors) +
theme_cowplot(font_size = 12) +
theme(legend.position = "none", panel.spacing = unit(0, "lines"),
plot.title = element_text(hjust = 0.5),
panel.background = element_rect(fill = NA, color = "black"),
plot.margin = margin(7, 7, 0, 7, "pt"),
strip.background = element_blank(),
strip.text = element_text(face = "bold"),
strip.text.y.left = element_text(angle = 0),
axis.title.x = element_blank(),
axis.ticks.x = element_blank(),
axis.text.x = element_blank() #隐藏
) +
ggtitle("Feature on x-axis with annotation") + ylab("Expression Level")
p2B:构建注释信息-基因分组信息
这里通路是随便写的,仅为示例,并不是该marker gene 在的通路。
# Create grouping info
df <- data.frame(x = levels(vln.dat.melt$gene),
group = c("A","A","B","B","B","B","B","C","C","C","D","D","D",
"D","D","D","D","D"),
stringsAsFactors = FALSE)
df$x <- factor(df$x, levels = levels(vln.dat.melt$gene))
df$group <- factor(df$group)
#可以修改 注释 展示的名字
levels(df$group) = c("ECM-receptor interaction", "PI3K-Akt signaling pathway",
"MAPK signaling pathway", "Cell adhesion molecules")
#设置颜色
color <- c("cyan", "pink", "green", "darkorange")
# guides() is used to specify some aesthetic parameters of legend key
p3 <- ggplot(df, aes(x = x, y = 1, fill = group)) + geom_tile() + theme_bw(base_size = 12) +
scale_fill_manual(values = my36colors) + scale_y_continuous(expand = c(0, 0)) +
guides(fill = guide_legend(direction = "vertical", label.position = "right",
title.theme = element_blank(), keyheight = 0.5, nrow = 2)) +
theme(legend.position = "bottom",
legend.justification = "left",
legend.margin = margin(0,0,0,0),
legend.box.margin = margin(-10,5,0,0),
panel.spacing = unit(0, "lines"),
panel.background = element_blank(),
panel.border = element_blank(),
plot.background = element_blank(),
plot.margin = margin(0, 7, 7, 7, "pt"),
axis.text.x = element_text(angle = 90, hjust = 1, vjust = 0.5, color = "black"),
axis.title.y = element_blank(),
axis.ticks.y = element_blank(),
axis.text.y = element_blank()) + xlab("Feature")
p3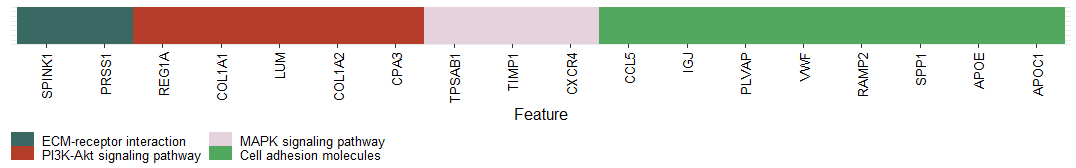
C:拼图收工
# Use plot_grid to join plots
plot_grid(p2, p3, ncol = 1, rel_heights = c(0.78, 0.22), align = "v", axis = "lr")
参考资料:
https://github.com/ycl6/StackedVlnPlot