Seurat V5|当单细胞进入百万细胞时代,BPCell 给出一种“解”决参考
Seurat V5|当单细胞进入百万细胞时代,BPCell 给出一种“解”决参考

现在单细胞的样本量和数据量都在逐渐增多,大有百万之势,如果用电脑分析就会显的吃力。这时候Seurat V5通过调用BPCells 也许能给出一个解决方案。
一 R包,数据准备
1,准备BPCells等R包
如果因网络问题出现报错的话,可以在github上下载BPCells的zip文件,然后本地安装 。之后缺少什么包就安装什么包 。
#devtools::install_github(c("bnprks/BPCells"))
library(Seurat)
library(BPCells)
library(ggplot2)
# needs to be set for large dataset analysis
options(future.globals.maxSize = 1e9)2,准备数据
使用https://cf.10xgenomics.com/samples/cell-exp/1.3.0/1M_neurons/1M_neurons_filtered_gene_bc_matrices_h5.h5中下载的h5数据进行示例 ,细胞数130万 。
##数据读取
brain.data <- open_matrix_10x_hdf5(path = "1M_neurons_filtered_gene_bc_matrices_h5.h5")
##输出表达矩阵到本地
write_matrix_dir(mat = brain.data,dir = "brain_counts")会在当前目录下根据write_matrix_dir生产一个对应名字的文件夹(brain_counts),内含有很多文件用于后续读取。
#查看数据
brain.mat <- open_matrix_dir(dir = "brain_counts")
brain.mat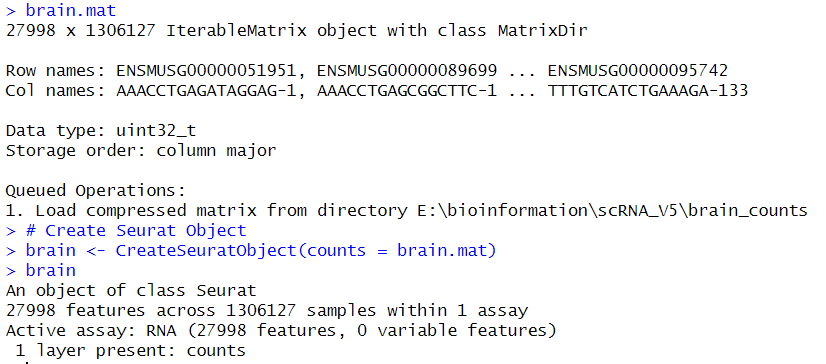
直接读取10X的三个文件会报错。
二 标准分析
1,创建Seurat 对象
根据前面读取的矩阵构建object对象,进行HVG之前的标准分析
# Create Seurat Object
pbmc <- CreateSeuratObject(counts = mat_raw)
pbmc
##标准化数据
brain <- NormalizeData(brain)
##识别高变基因
brain <- FindVariableFeatures(brain)head(brain)
orig.ident nCount_RNA nFeature_RNA
AAACCTGAGATAGGAG-1 SeuratProject 4046 1807
AAACCTGAGCGGCTTC-1 SeuratProject 2087 1249
AAACCTGAGGAATCGC-1 SeuratProject 4654 2206
AAACCTGAGGACACCA-1 SeuratProject 3193 1655
AAACCTGAGGCCCGTT-1 SeuratProject 8444 3326
AAACCTGAGTCCGGTC-1 SeuratProject 11178 38662,抽取细胞
使用SketchData函数抽取细胞,ncells参数指定抽取多少细胞到内存中进行分析,注意指定assay名字,方便后续分析。其他细胞仍然可以硬盘访问 。
##抓取2w细胞载入到内存中进行分析
brain <- SketchData(object = brain,
ncells = 20000,
method = "LeverageScore",
sketched.assay = "sketch")
brain
An object of class Seurat
55996 features across 1306127 samples within 2 assays
Active assay: sketch (27998 features, 2000 variable features)
2 layers present: counts, data
1 other assay present: RNA
dim(brain@assays$sketch$counts)
#[1] 27998 20000可以看到已完成2W细胞的抽取,接下来进行后续分析。
3,Sketch细胞标准分析
将默认对象切换到sketch(内存中的2w细胞),然后进行标准分析
# 默认对象切换到sketch(内存中的2w细胞)
DefaultAssay(brain) <- "sketch"
brain <- FindVariableFeatures(brain)
brain <- ScaleData(brain)
brain <- RunPCA(brain)
brain <- FindNeighbors(brain, dims = 1:50)
brain <- FindClusters(brain, resolution = 2)
brain <- RunUMAP(brain, dims = 1:50, return.model = T)
brain
除了上述sketch抽取的2W外,其他的cluster信息(sketch_snn_res.2)均为NA 。
(2)基于2W细胞进行可视化
##绘制umap图(2w细胞)
p1 <- DimPlot(brain, label = T, label.size = 3, reduction = "umap") + NoLegend()
p1
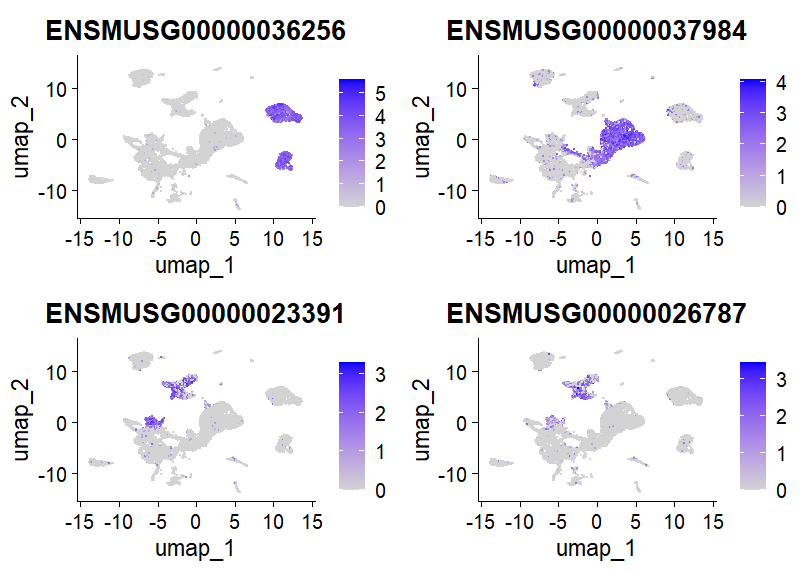
p2 <- FeaturePlot(object = brain,
features = c("ENSMUSG00000036256",
"ENSMUSG00000037984",
"ENSMUSG00000023391",
"ENSMUSG00000026787"),
ncol = 2)
p2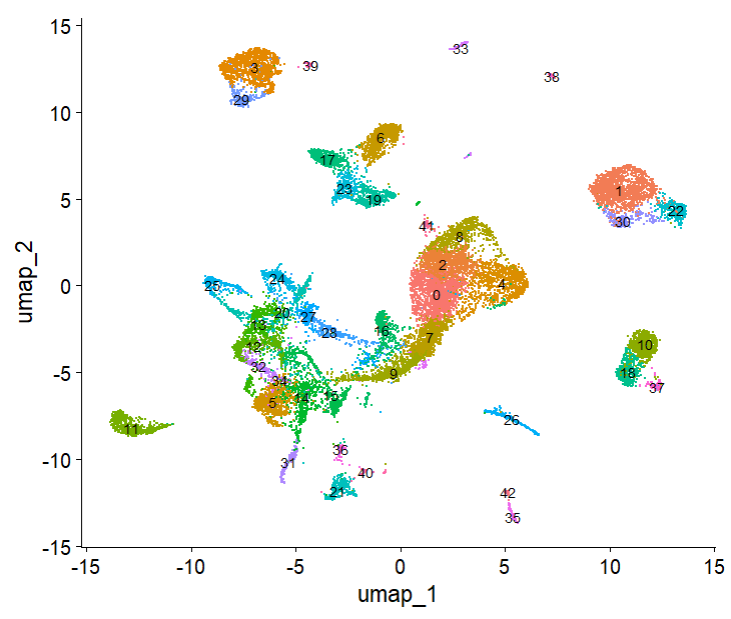
4 扩展至全部细胞
使用ProjectData函数将sketch得到的cluster和reduction信息扩展至全部百万细胞。
扩展后的降维,聚类结果存放在含有full的结果中,如pca.full, full.umap和cluster_full 。
##扩展(从2w细胞扩展到130w细胞)
brain <- ProjectData(object = brain,
assay = "RNA",
full.reduction = "pca.full",
sketched.assay = "sketch",
sketched.reduction = "pca",
umap.model = "umap",
dims = 1:50,
refdata = list(cluster_full = "seurat_clusters"))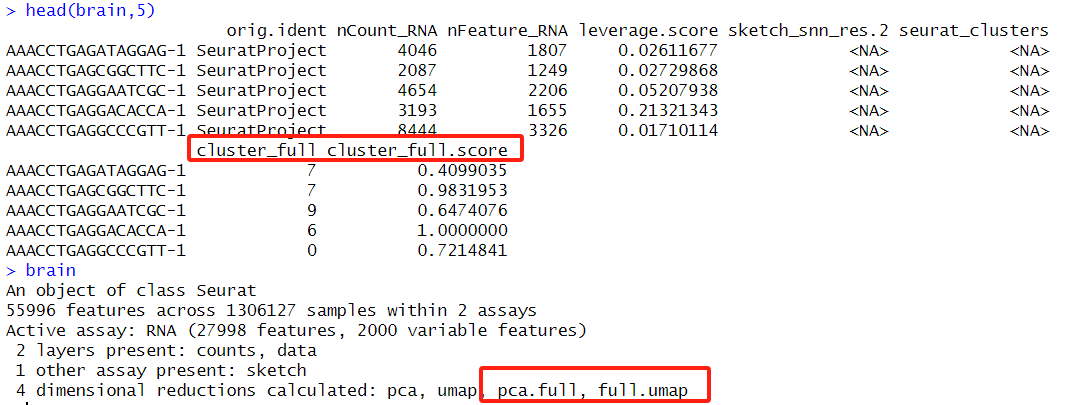
新增的cluster_full即为根据sketch的cluster预测出来的全部细胞的cluster结果 ,cluster_full.score为预测的得分。
更多细节可以通过View(TransferSketchLabels)查看

(2)基于全部细胞可视化
##默认对象切换到RNA(全部数据集)
DefaultAssay(brain) <- "RNA"
p3 <- DimPlot(brain, label = T,
label.size = 3,
reduction = "full.umap",
group.by = "cluster_full",
alpha = 0.1) + NoLegend()
p1 + p3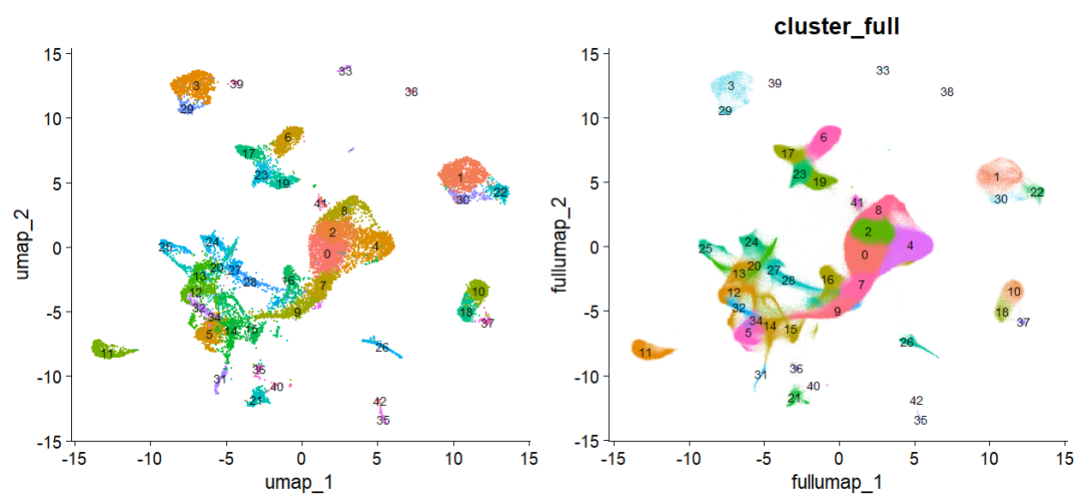
(3)以两个基因为例,比较sketch 和 全部的结果
DefaultAssay(brain) <- "sketch"
x1 <- FeaturePlot(brain, features = c("ENSMUSG00000036256", "ENSMUSG00000036887") )
DefaultAssay(brain) <- "RNA"
x2 <- FeaturePlot(brain, features = c("ENSMUSG00000036256", "ENSMUSG00000036887") )
p4 <- x1 / x2
p4
可以看到推断至全部的一致性还是可以的。
至此就借用该种方式完成了百万级别细胞的单细胞分析,当然内存足够的可以直接用全部细胞走正常的流程。
后续也可以进行亚群分析,提取感兴趣的亚群,根据细胞数选择继续Sketch 或者 进行正常的分析(此处不展示,详见参考资料)。
参考资料:
https://bnprks.github.io/BPCells/articles/pbmc3k.html
https://satijalab.org/seurat/articles/seurat5_sketch_analysis