Seurat_V5|单细胞转录组 + 蛋白,WNN方法分析单细胞多模态数据
Seurat_V5|单细胞转录组 + 蛋白,WNN方法分析单细胞多模态数据

前面Seurat V5|一个函数就能解决多种去批次方法,按需尝试提到V5的升级部分(https://satijalab.org/seurat/articles/get_started_v5_new)主要体现在4个方面,本次介绍 Seurat V5 的WNN方法分析单细胞多模态数据,本文以转录组+蛋白组数据为例。
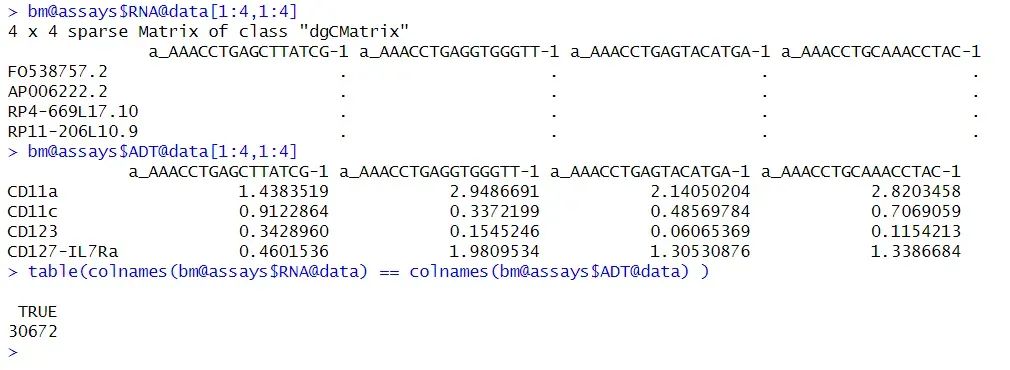
使用Stuart*, Butler* et al, Cell 2019提供的CITEseq数据作为示例,含有30,672 scRNA-seq 和 25 antibodies数据。
一 载入R包,数据
使用SeuratData中的bmcite数据示例,展示CITEseq数据中的单细胞转录组和蛋白数据的结合 。
如果代码直接安装bmcite有问题的小伙伴可以在网页中输入http://seurat.nygenome.org/src/contrib/bmcite.SeuratData_0.3.0.tar.gz 直接下载tar文件,然后本地安装。
library(Seurat)
library(SeuratData)
library(cowplot)
library(dplyr)
#InstallData("bmcite")
install.packages('./bmcite.SeuratData_0.3.0.tar.gz', repos = NULL, type = "source")
bm <- LoadData(ds = "bmcite")
bm
An object of class Seurat
17034 features across 30672 samples within 2 assays
Active assay: RNA (17009 features, 2000 variable features)
2 layers present: counts, data
1 other assay present: ADT
1 dimensional reduction calculated: spca可以看到对象中有RNA 和 ADT 2个assay。确认下RNA和ADT的cell barcode 是否一致
二 WNN 模态数据处理
1,RNA标准分析
先指定默认assay为RNA,然后进行至PCA的标准分析
DefaultAssay(bm) <- 'RNA'
bm <- NormalizeData(bm) %>%
FindVariableFeatures() %>%
ScaleData() %>%
RunPCA()
bm
An object of class Seurat
17034 features across 30672 samples within 2 assays
Active assay: RNA (17009 features, 2000 variable features)
3 layers present: counts, data, scale.data
1 other assay present: ADT
2 dimensional reductions calculated: spca, pca2, ADT标准分析指定默认assay为ADT,然后标准分析
DefaultAssay(bm) <- 'ADT'
# we will use all ADT features for dimensional reduction
# we set a dimensional reduction name to avoid overwriting the
VariableFeatures(bm) <- rownames(bm[["ADT"]])
bm <- NormalizeData(bm, normalization.method = 'CLR', margin = 2) %>%
ScaleData() %>%
RunPCA(reduction.name = 'apca')
bm
An object of class Seurat
17034 features across 30672 samples within 2 assays
Active assay: ADT (25 features, 25 variable features)
3 layers present: counts, data, scale.data
1 other assay present: RNA
3 dimensional reductions calculated: spca, pca, apca注:这里使用所有的ADT特征进行降维,此外注意设置reduction.name,以避免覆盖 。 3,WNN
对于每个细胞,我们根据RNA和蛋白质相似性的加权组合识别数据集中的多模态邻居,并将结果存储在neighbors插槽中,注意reduction.list中的pca 和 apca 要和前面单独分析时定义的名字一致
# Identify multimodal neighbors. These will be stored in the neighbors slot,
# and can be accessed using bm[['weighted.nn']]
# The WNN graph can be accessed at bm[["wknn"]],
# and the SNN graph used for clustering at bm[["wsnn"]]
# Cell-specific modality weights can be accessed at bm$RNA.weight
bm <- FindMultiModalNeighbors(
bm,
reduction.list = list("pca", "apca"),
dims.list = list(1:30, 1:18),
modality.weight.name = "RNA.weight"
)
bm
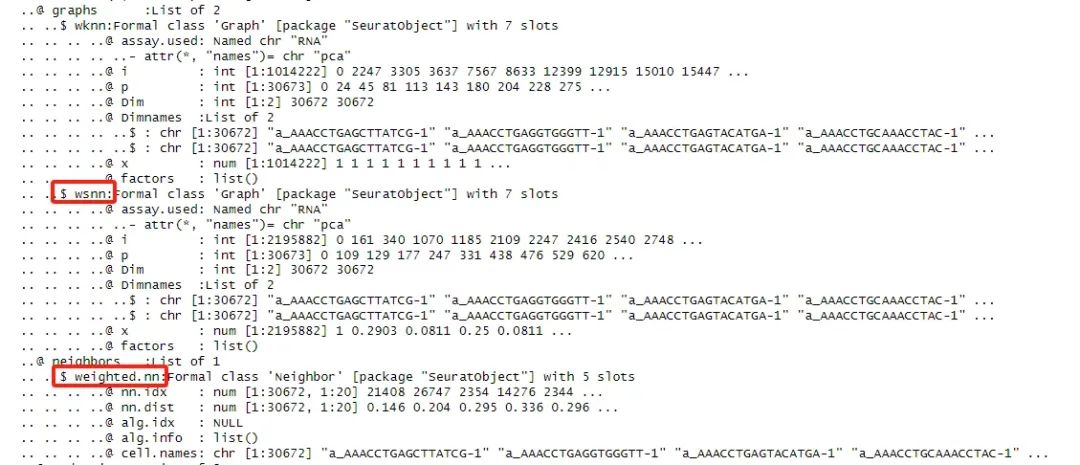

这里修改modality.weight.name 可能不起作用,通过View(FindMultiModalNeighbors) 查看函数内容发现名字是和assay有关的,没啥影响。
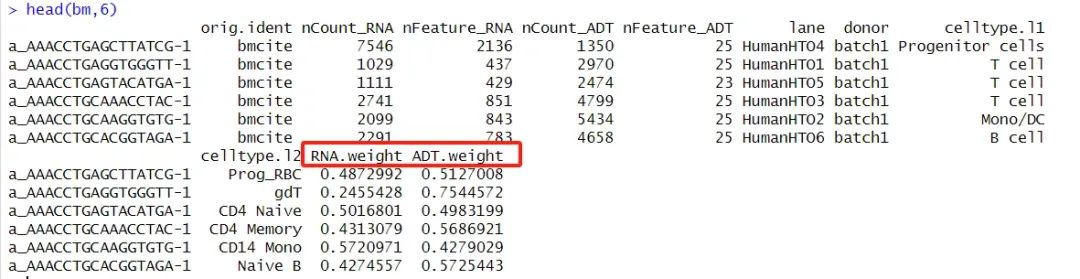
然后就可以进行后续的降维 聚类分析了。
4,降维聚类
上述WNN方法完成数据合并后,分别可以基于(1)转录组 (2)蛋白组 (3)以及结合权重的数据 进行UMAP 可视化分析。(1)基于 RNA + protein 的结合数据 注意这里不是assay,而是nn.name 参数指定,当然要注意reduction.name 。
bm <- RunUMAP(bm, nn.name = "weighted.nn",
reduction.name = "wnn.umap",
reduction.key = "wnnUMAP_")
bm <- FindClusters(bm,
graph.name = "wsnn",
algorithm = 3,
resolution = 2, verbose = FALSE)
bm
An object of class Seurat
17034 features across 30672 samples within 2 assays
Active assay: ADT (25 features, 25 variable features)
3 layers present: counts, data, scale.data
1 other assay present: RNA
4 dimensional reductions calculated: spca, pca, apca, wnn.umap
p1 <- DimPlot(bm, reduction = 'wnn.umap',
label = TRUE,
repel = TRUE, label.size = 2.5) +
NoLegend()
p2 <- DimPlot(bm, reduction = 'wnn.umap',
group.by = 'celltype.l2',
label = TRUE, repel = TRUE,
label.size = 2.5) +
NoLegend()
p1 + p2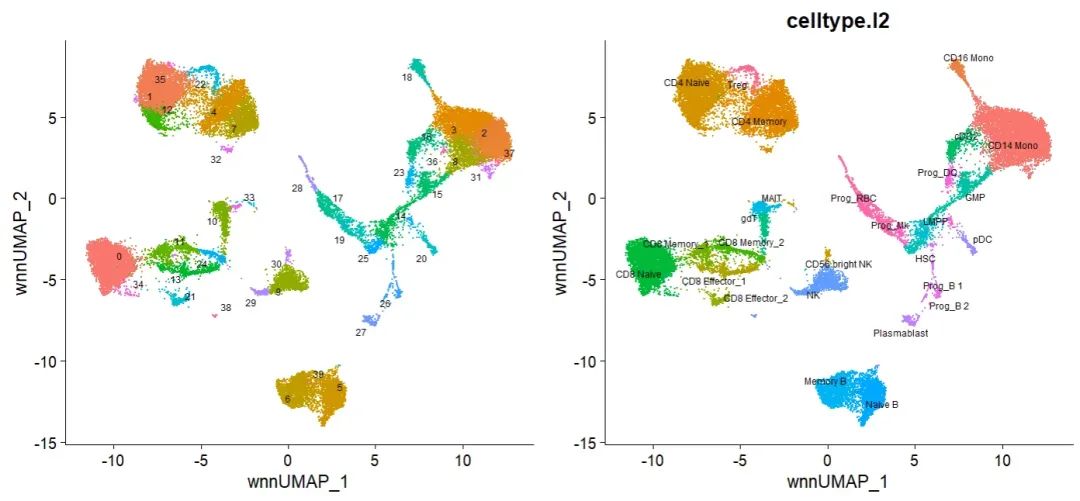
(2)单独基于 RNA 或 protein 数据
bm <- RunUMAP(bm, reduction = 'pca', dims = 1:30, assay = 'RNA',
reduction.name = 'rna.umap', reduction.key = 'rnaUMAP_')
p3 <- DimPlot(bm, reduction = 'rna.umap', group.by = 'celltype.l2', label = TRUE,
repel = TRUE, label.size = 2.5) + NoLegend()
bm <- RunUMAP(bm, reduction = 'apca', dims = 1:18, assay = 'ADT',
reduction.name = 'adt.umap', reduction.key = 'adtUMAP_')
p4 <- DimPlot(bm, reduction = 'adt.umap', group.by = 'celltype.l2', label = TRUE,
repel = TRUE, label.size = 2.5) + NoLegend()
p3 + p4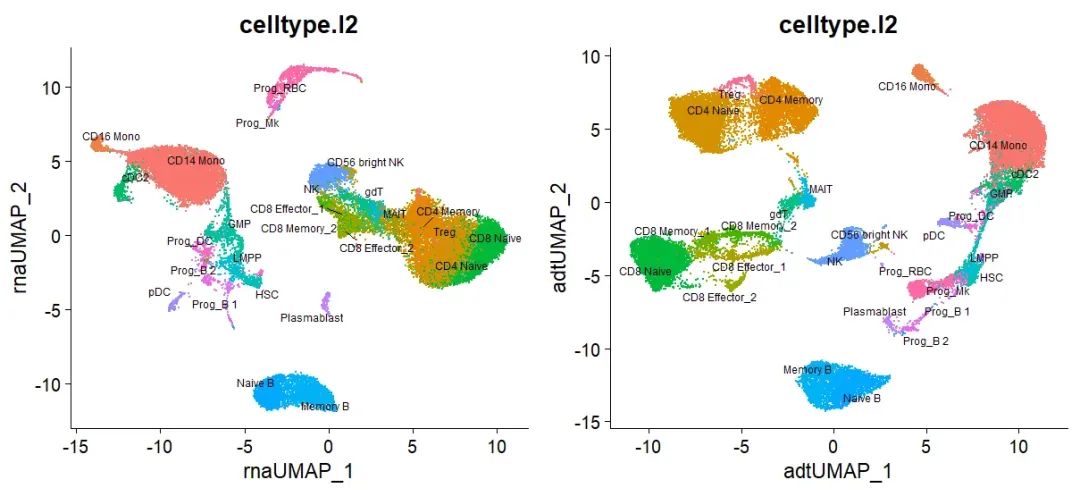
(3)经典marker 可视化 查看典型marker基因和蛋白在多模态整合后的UMAP上的表达,为注释提供参考
p5 <- FeaturePlot(bm, features = c("adt_CD45RA","adt_CD16","adt_CD161"),
reduction = 'wnn.umap', max.cutoff = 2,
cols = c("lightgrey","darkgreen"), ncol = 3)
p6 <- FeaturePlot(bm, features = c("rna_TRDC","rna_MPO","rna_AVP"),
reduction = 'wnn.umap', max.cutoff = 3, ncol = 3)
p5 / p6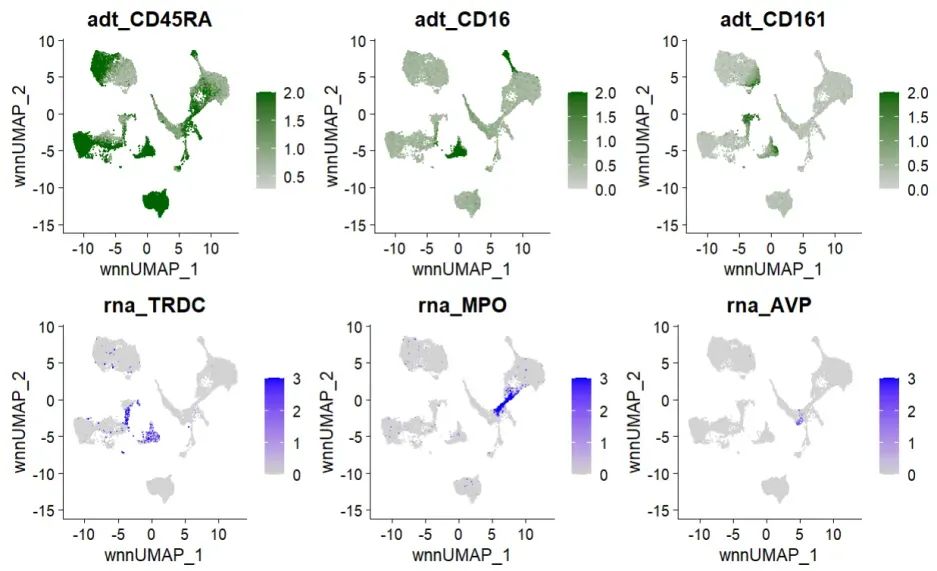
ADT中含有的蛋白数量和种类不一致,综合考虑一些经典marker的蛋白 和 基因,可以更好的进行注释 以及 对注释进行验证。