scTCR+scRNA | APackOfTheClones - umap坐标下球形展示celltype的clone size
scTCR+scRNA | APackOfTheClones - umap坐标下球形展示celltype的clone size

APackOfTheClones 是2021年的Single-cell analysis pinpoints distinct populations of cytotoxic CD4+ T cells and an IL-10+CD109+ TH2 cell population in nasal polyps文献中的一种克隆型计数分布的可视化方式。通过每个球对应于给定cluster中的一个克隆型,球的大小与celltype中该克隆的细胞数量成正比,且对应的位置与UMAP图近似。

一 载入R包,数据
首先下载,载入R包APackOfTheClones 。仍然使用前面免疫组库相关推文的数据,分别(1)使用Seurat的标准流程处理scRNA 和(2)使用 scRepertoire 的标准流程处理 scTCR 数据 (3)然后结合scRNA 和 scTCR的数据,详细的分析过程见 单细胞免疫组库VDJ| 从零开始scRepertoire分析,解决真实场景中可能的问题
#install.packages("APackOfTheClones")
library(APackOfTheClones)
library(Seurat)
library(scRepertoire)
library(tidyverse)
library(ggsci) #获取期刊经典颜色
library(scales) #展示颜色
load("combined_seurat_T.RData")
head(seurat_T,2)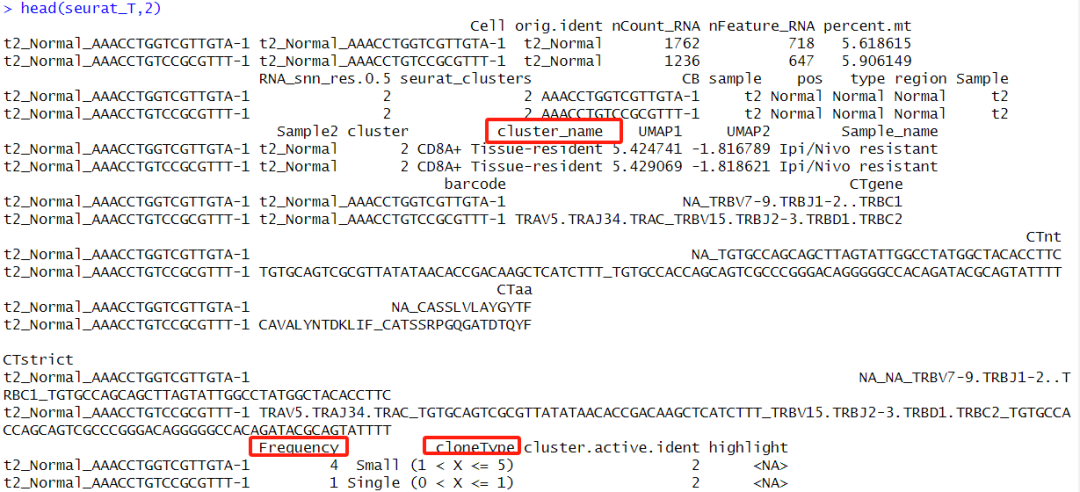
二 scRepertoire可视化
使用常规方式在umap图中展示cloneType的信息
colorblind_vector <- colorRampPalette(rev(c("#0D0887FF", "#47039FFF","#7301A8FF", "#9C179EFF",
"#BD3786FF", "#D8576BFF","#ED7953FF","#FA9E3BFF",
"#FDC926FF", "#F0F921FF")))
DimPlot(seurat_T, group.by = "cloneType",label = F) +
scale_color_manual(values=colorblind_vector(5)) +
theme(plot.title = element_blank())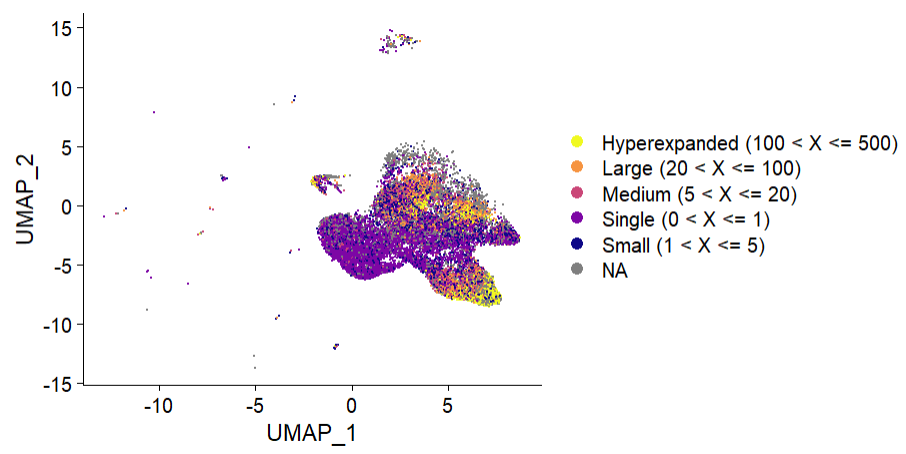
前文提取出T细胞后,未做分析处理。这里为了后续的展示,先进行T细胞亚群的标准流程
sce.T <- NormalizeData(seurat_T)
sce.T <- FindVariableFeatures(sce.T, selection.method = "vst", nfeatures = 2000)
sce.T <- ScaleData(sce.T)
sce.T <- RunPCA(sce.T, npcs = 20)
#标准流程,参数不变
sce.T <- sce.T %>%
RunUMAP(dims = 1:20) %>%
FindNeighbors(dims = 1:20) %>%
FindClusters(resolution = c(0.05, 0.1))
DimPlot(sce.T, group.by = "RNA_snn_res.0.1",label = F)
T细胞的亚群注释,本推文重点不在注释,因此这里就根据meta的信息简单注释下 ,
Idents(sce.T) <- "RNA_snn_res.0.1"
sce.T <- RenameIdents(sce.T,
"0"="CD8A+ Exhausted",
"1"="CD4+ Naive",
"2"="CD8A+ Tissue-resident",
"3"= "CD8A+ NK-like",
"4"= "CD4+ Effector",
"5"= "CD8A+ NK-like",
"6"= "CD8A+ NK-like",
"7"= "CD4+ Activated IEG",
"8"= "CD8A+ Proliferating",
"9"= "CD8A+ NK-like" ,
"10"="CD8A+ NK-like"
)
sce.T@meta.data$celltype <- Idents(sce.T)
DimPlot(sce.T, reduction = 'umap',
label = T, pt.size = 0.5)
三 APackOfTheClones
通过以上过程得到了scTCR 和 scRNA数据结合在一起的sce.T文件,接下来使用APackOfTheClones进行可视化展示。通过各个cluster/celltype中圈的大小代表clone size的大小 。
3.1 默认参数
#不管用
Idents(sce.T) <- "celltype"
p0 <- vizAPOTC(sce.T, verbose = FALSE)
p0
上述结果表明"CD8A+ Exhausted" 和 "CD8A+ NK-like" 细胞类型中clone 较多 。
可以看到即使设置了Idents 还是按照cluster 着色的,通过View(vizAPOTC) 查看函数也未发现可以调整的参数。
注1:后续根据猜测和测试验证,发现vizAPOTC函数会按照seurat_clusters列进行展示 ,暂时使用如下操作进行处理
# 备份原seurat_clusters结果
sce.T$seurat_clusters2 <- sce.T$seurat_clusters
# 将注释结果赋值给 seurat_clusters
sce.T$seurat_clusters <- sce.T$celltype3.2 图形美化
和期刊发表图相比,可以有以下几个地方可以进一步的调整 :(1)标签Clone Sizes 的位置以及数值,(2)线型,大小 (3)按照细胞类型着色 。
# vizAPOTC 调整
p1 <- vizAPOTC(sce.T,
legend_sizes = c(1,10,50), # 指定Clone Sizes的数值
add_legend_background = T , #是否添加灰色背景
order_clones = TRUE, #
legend_text_size = 3, #legend 标签大小
legend_position = "top right" , #legend的位置 ,可以根据umap的方位进行调整
try_place = T,
res = 360L,
linetype = "blank",
use_default_theme = TRUE,
retain_axis_scales = FALSE,
show_labels = F, # 图中不展示标签
label_size = 3, #
verbose = FALSE)
p1
#调整颜色
p11 <- p1 + scale_fill_nejm()
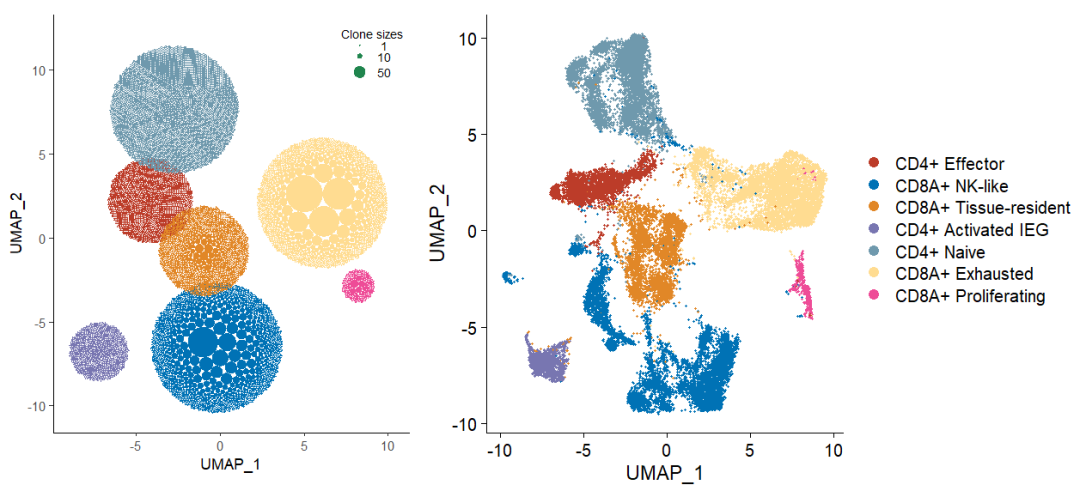
p11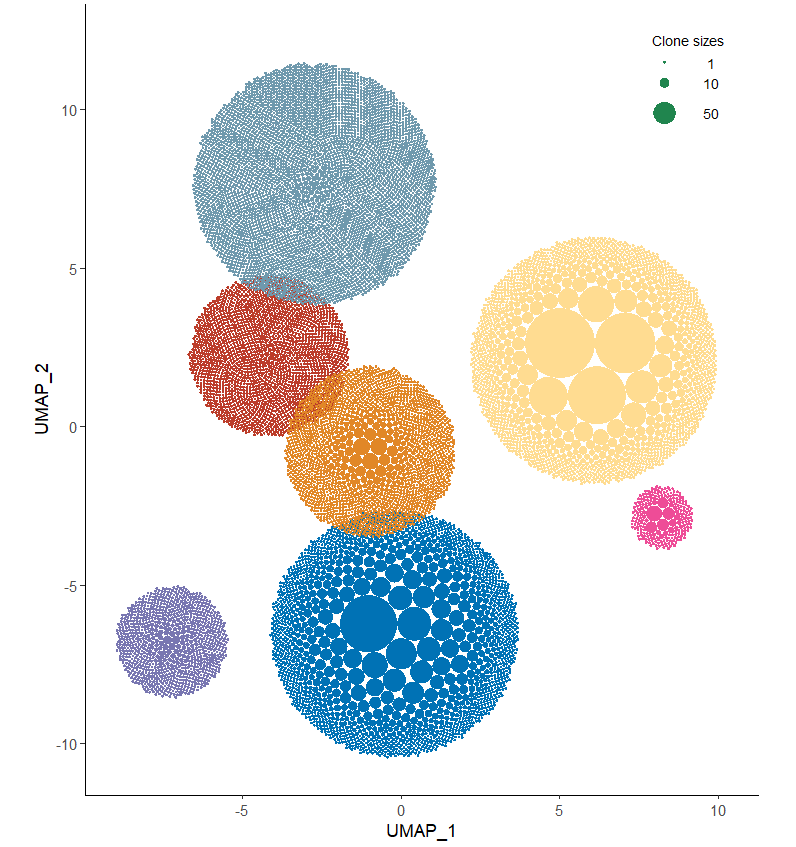
可以看到将注释结果celltype赋值给 seurat_clusters后,现在是按照细胞类型进行着色(也许有更好的方法)。
注2:scale_fill_nejm 颜色顺序和 seurat_clusters的factor顺序不一致 。
且#20854EFF的绿色莫名其妙的变成了legend的颜色。
注3:??vizAPOTC 的帮助页有很多的参数,这里只是展示了几个常用的,更多详细说明见官网。
3.3 调整scRNA umap颜色
上面的注2提到颜色顺序的问题,由于vizAPOTC函数包装的比较“严”,这里通过重新定义factor的顺序完成scRNA的颜色修改,使之与clone的颜色一致 。(也许有更好的方法)
(1)查看scale_fill_nejm的颜色
# 输出颜色编码
pal_nejm("default")(8)
#[1] "#BC3C29FF" "#0072B5FF" "#E18727FF" "#20854EFF" "#7876B1FF" "#6F99ADFF" "#FFDC91FF" "#EE4C97FF"
# 展示颜色
show_col(pal_nejm("default")(8))
(2)修改factor顺序指定颜色
结合vizAPOTC的颜色 和 nejm的颜色代码,重新定义levels 或者 对应修改cols的顺序,总之对应好即可。
Idents(sce.T) <- "seurat_clusters"
sce.T$seurat_clusters <- factor(sce.T$seurat_clusters ,
levels = c("CD4+ Effector" , "CD8A+ NK-like" ,
"CD8A+ Tissue-resident" ,
"CD4+ Activated IEG" ,
"CD4+ Naive" ,
"CD8A+ Exhausted" ,
"CD8A+ Proliferating"))
p2 <- DimPlot(sce.T, reduction = 'umap',
cols = c("#BC3C29FF", "#0072B5FF" ,"#E18727FF",
"#7876B1FF" ,"#6F99ADFF", "#FFDC91FF","#EE4C97FF"),
label = F, pt.size = 0.5 )
p2
这样就完成了颜色的修改以及对应,可以放到文章中了。
参考资料
[1]https://qile0317.github.io/APackOfTheClones/articles/APackOfTheClones.html
[2]Single-cell analysis pinpoints distinct populations of cytotoxic CD4+ T cells and an IL-10+CD109+TH2??2cell population in nasal polyps
[3]Recombinant multimeric dog allergen prevents airway hyperresponsiveness in a model of asthma marked by vigorousTH2??2andTH17??17cell responses