小站工具2.0|单基因生存期曲线图+ROC,重点是根据批量生成的单因素COX与AUC结果筛选~回复:SUV,可获得测试文件!
小站工具2.0|单基因生存期曲线图+ROC,重点是根据批量生成的单因素COX与AUC结果筛选~回复:SUV,可获得测试文件!
能用鼠标解决的事,绝对不用代码
做一些生信美图
你需要这个工具 Easy Tools v2.0
下面网址是工具入口:
www.chrislifescience.club:3838/R/AnnoE2/
2.0重磅更新:可根据AUC与单因素COX结果筛选的生存曲线+ROC图
这里大大的感谢:Daying!
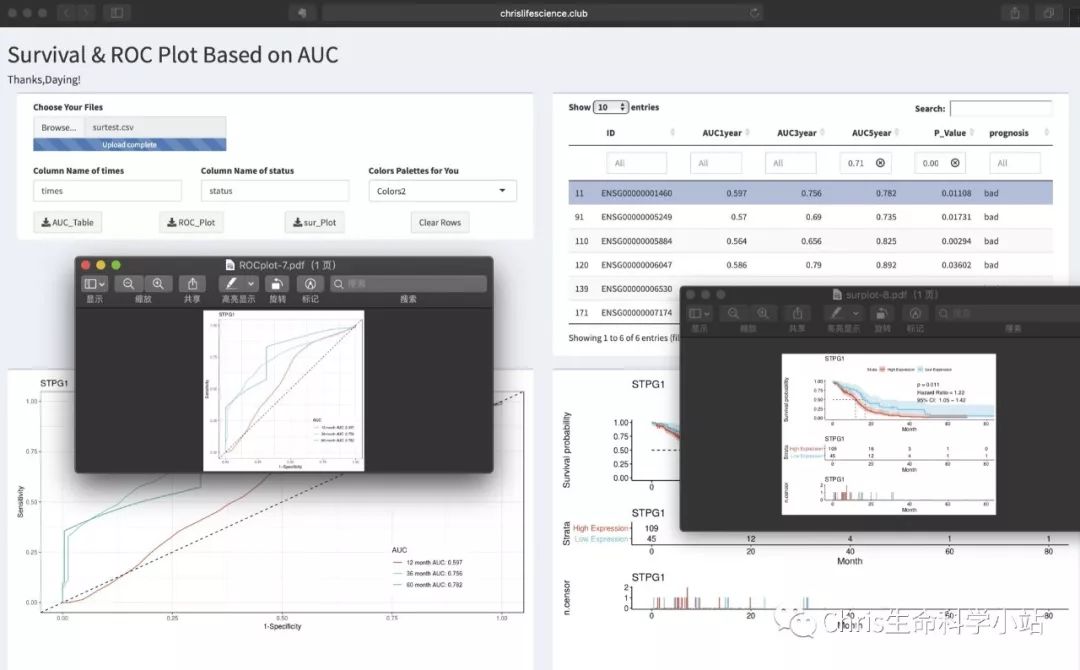
1、数据准备:
1)格式csv。其中包括生存期数据(时间+事件状态)例如这样:

2、运行
2)输入进去以后,你需要等待~~因为第一步是批量并行计算单因素COX+AUC,这个过程站长测试,200基因x150样本,至少要1分钟。一般要等1分钟,结果出来以后右上角那个表格会有变化。
3)右上角的表格可以进行,各列筛选,比如5年生存期AUC>0.7 & 单因素COX p<0.05 & prognosis is bad。
4)选择你要的那个基因,以后下面会自动生成ROC与生存期曲线。
3、输出
5)当然可以调色
6)当然可以下载,了解站长的应该知道怎么下。
同学们看黑板,这里要重点说明一下!!!!!
批量COX+AUC真的很吃机!
站长服务器cpu是10核20线程的单cpu,试过跑2000基因就要3分钟了,推测跑40000个基因要跑1个小时。
求诸位放过站长的服务器和家里的电费。
基因筛选一下在跑,最好是200个基因x200以内个样本。
如果跑的过程中,出现
画面不动的情况,千万别盯着,去刷刷朋友圈,看看站长的教程。
如果没有结果过了五分钟还没有出现,要淡定,去筛筛自己的基因,缩小一下范围。再来一次!
可能有同学要问,站长加一个cpu不就解决问题了!
嗯,这个同学很聪明!
加个cpu大概需要5000RMB,
但是,因为钱都给娃买奶粉了,真的加不起了!
如果真的想更好的体验这个工具,只能靠大家自己了。
站长开了赞赏通道,网站来源的赞赏,都用于升级服务器使用。
升级了也能为大家更好的服务。
在这里谢谢大家了~
ps这几个功能写完TCGA的课程可以更新了,
如何配合网站工具去做TCGA转录组分析呢?课程会以直播的形式授课,时间会在公众号通知,敬请期待吧!
1.6 重磅更新:GSEA功能上线!
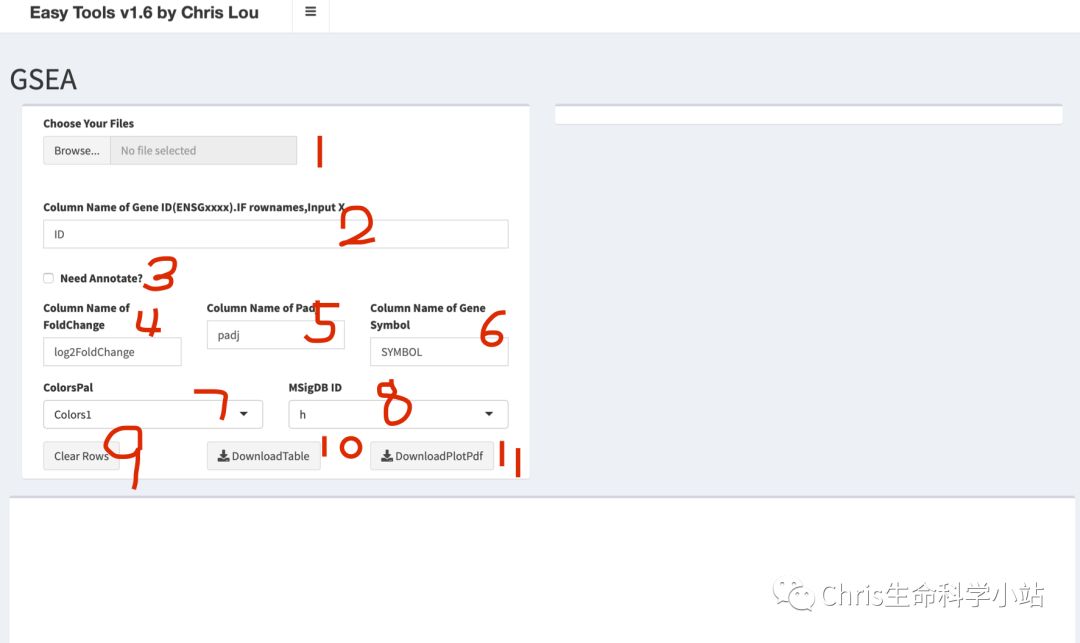
1、数据准备:
1)格式csv,在上1的位置输入。
2)如果基因名是ENSGxxxx的不要担心,在2的位置ENSG所在那列的名字。在3的地方勾选。下面的456就不要改动了。
3)如果是芯片数据,或者自己DIY的数据,数据中至少应该包括:倍数列,p值列,基因名列。分别在图中4、5、6填入。注意!这时在3的地方不要勾选。
2、出表出图:
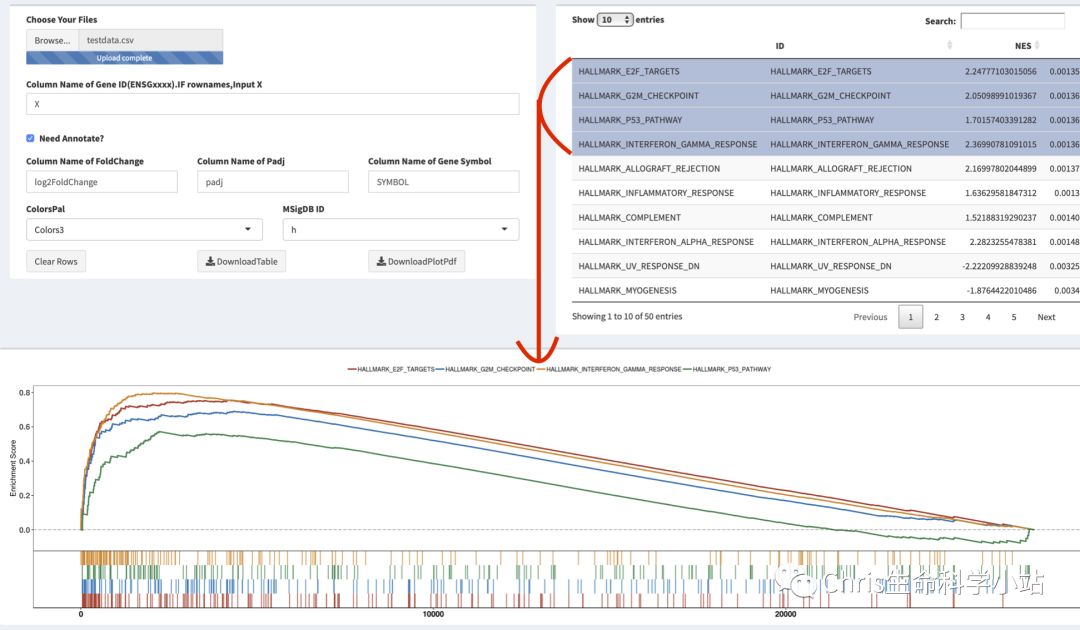
1)图中右上角的表格是GSEA的结果,10的位置可以下载。
2) 下面的图是GSEA的结果图,可以在表格中选择想画图的基因集ID。这里可以多选(最多10个),11的位置下载的是GESA的PDF图像。
3)7的位置准备了三个配色方案可选择
4)在8的位置选择Geneset,c1站长从来没用过,直接去掉。选择c2的时候要等待15秒右下角有进度条。
站长的感悟
我们成长在一个非常好的时代,在这个时代,掌握一门技术,学习某些知识,因为互联网的存在而变得容易。
站长从构思到实现这样一个功能仅用了不到48小时,这对于一个临床医生来说,真的算是个奇迹了!
回想起来,这都得益于前辈们铺的路,比如徐洲更的shiny教程,比如Y叔的神包(clusterProfiler),再比如小丫画图的众筹模式,再比如Daying的代码等等,让一个只会回车的小白走向了开发工具的道路,在这里再次感谢你们!
以往更新的内容
1.5更新内容
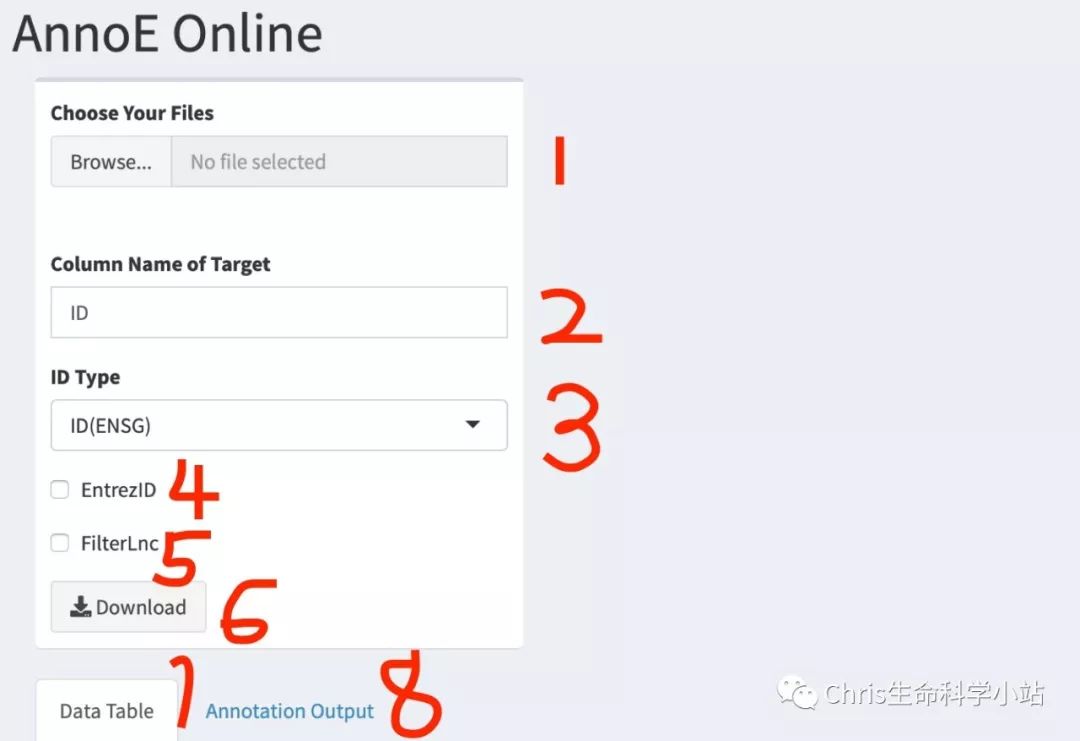
1、在1的位置输入,表格要求csv格式。在7的位置可以查看输入表格的结果。
2、在2的位置填写注释那列的列名。如果注释列为行名(rownames),注意这里需要填X。
3、在3的位置选择用什么注释,可以是ENSG,基因名或EntrezID。
4、如果需要EntrezID的内容在4这里打钩,如果3的位置选EntrezID默认打钩。
5、如果需要提取lncRNA(long noncoding RNA)在5这打钩
6、在8的位置会显示注释位置。
7、在6的位置点击下载注释以后的表格。
火山图的使用方法:
一、输入的表格要求:
1、格式为csv!!!
至少要有三列:分别是
基因名(Gene Symbol),倍数(log2FlodChange),p值(padj),在右上那几个列名中分别填写自己表格中各列的名字。
二、如果geneID是ENSG,内置注释功能,点选Need Annotation输入ENSG列名。
1.3更新内容:
去掉了ployly渲染,那个渲染交互真的很好。但是有两个缺点一个是不能输出矢量pdf图,另外一个就是太卡了。所以站长舍弃了ployly,自己操刀实现了几个经常用到的交互功能。
1.火山图加上自己配色选项。之前站长精选配色依然可以选择,如果想自己DIY,请选择YouLike,然后在调色板选颜色,火山图会实时更新。
2.升级指示基因功能。可以实现单选多选,并与表格交互。你想选哪个基因,在右边表格点一下。左边图就会更新。
ps出现红色的提示不要担心,记得把Select Gene那个勾上。
3.优化输出。图片可以直出矢量pdf,不用担心图像因为页面大小变形,输出的pdf是正常的,这样在手机上也可以使用。
下面这个是使用视频教程。
One More Thing.......
配合AnnoE的功能,可以实现提取LncRNA后做火山图,具体操作看下面:
1.2更新内容:
如果想选某个基因,在2的位置勾选,在1的位置填写准确填写基因SYMBOL(如果不知道准确信息,可以到Geneinfo中去查询)
3的位置是下载火山图pdf的位置,图生成完点击就好!

当然这个工具还可以实现
1、基因信息查询

之前需要输入准确的基因名字,现在可以实现一丢丢模糊搜索:
不必记住全名,输入几个字母,就能返回结果,可以在下面列表查看结果。
如果是ENSG开头的,还是要输入准确的~
这个工具是跨平台的,
手机,平板,电脑都可以,
只要在浏览器输入下面这个网址:
www.chrislifescience.club:3838/R/AnnoE2/
以后会更新的功能: