空转数据分析之特异性生态位基因依赖的细胞通讯
原创空转数据分析之特异性生态位基因依赖的细胞通讯
原创
作者,Evil Genius
周二了,又是催相亲的一天,大概率又要见一个奇葩女。
空间的邻域通讯汇总过了,文章在空间邻域通讯分析大汇总
空间的分子niche,也分享过了,文章在空间转录组学数据分析细胞邻域依赖的基因表达(分子邻域),对应的是空间邻域分子矩阵。
那么在实际的分析中,我们需要二者结合,也就是Niche-DE与Niche-LR要联合分析。
适用的空间平台:CosMx SMI、Slide-seq和Xenium、Visium、CODEX以及最新的HD。
联合的核心也很简单,一方面需要识别细胞类型特异性生态位相关基因,另一方面结合特异性生态位相关基因的所对应的LR或者转录因子,全方位认知生态位的形成。这是一种揭示niche差异基因表达模式基础的配体受体信号机制的方法。
我们今天参考的文献在Niche-DE: niche-differential gene expression analysis in spatial transcriptomics data identifies context-dependent cell-cell interactions,2024年1月发表于Genome Biology。
组织内的细胞必须不断协同工作以适应不断变化的环境。为了协调这些变化,使用了诸如旁分泌信号的通信机制。在旁分泌信号中,细胞释放化学物质,如激素、神经递质和生长因子,这些化学物质与邻近细胞上的特定蛋白质受体结合,引起这些细胞功能的变化。因此,细胞中的特定基因表达程序可能是由其生态位中其他细胞的活动驱动的。
空间转录组学(ST)技术正在迅速成熟,能够在转录组尺度上对基因表达进行高分辨率的原位测量。这些技术包括Merfish、SeqFISH+、Visium、Slide-seq和Stereo-seq(当然还有最新的HD)。为了挖掘这些数据,已经提出了用于图像分割、技术伪影去除、SPOT数据的SPOT反卷积、空间可变基因检测、邻域检测、细胞-细胞相互作用分析和其他分析的计算方法。特别是,检测空间可变基因,即在表达中显示明确空间模式的基因,已成为标准的分析步骤。空间可变基因可用于帮助组织病理学常规执行的任务,例如组织结构的可视化,并进一步识别具有不同空间定位的细胞类型。一旦在宏观水平上确定了细胞类型的分布,就会对局部的、细胞类型特异性的相互作用进行研究。通过设计,空间可变基因分析旨在识别基因表达的全局模式,而不是细胞类型之间的局部相互作用。例如,在癌症组织中,空间可变基因使我们能够区分癌症和正常组织区域;然而,我们需要识别和评估局部模式的重要性,如肿瘤和免疫细胞之间的生态位信号。
细胞之间的局部相互作用,如基于信号通路或细胞外基质的化学和生物力学重塑的相互作用,是高度细胞类型特异性的。因此,需要探索细胞类型特异性模型,以了解细胞基因表达程序对其局部微环境生态位的依赖性。在分析中,将细胞的生态位定义为其局部邻域的细胞类型组成,其中局部邻域的范围由kernel function决定。分析预计细胞的生态位会影响其基因表达谱,为此,需要一种新的统计方法,称为生态位差异表达(Niche-DE)分析。Niche-DE鉴定细胞类型特异性生态位相关基因,定义为与细胞类型特异性平均表达相比,在特定空间生态位背景下,特定细胞类型中的表达显著上调或下调的基因。
空间可变基因分析寻找与某些空间模式相关的表达模式的基因,而细胞-细胞相互作用分析寻找比随机机会更容易共定位的细胞类型。
与空间可变基因分析相比,Niche-DE表达分析的一个显著的特点是更容易在多个样本中识别生态位的变化。“巨噬细胞在靠近肿瘤细胞时上调特定基因X的表达”这样的说法可以在多个组织切片上应用和测试。因此,Niche-DE分析可以自然地整合来自多个组织载玻片和患者的数据,并具有纠正组织和载玻片批次效应的策略。
当Niche-DE表达基因被确定后,一个自然的后续问题是是否有一种已知的细胞-细胞信号机制导致了它们的上调或下调。为了实现这一目标,则需要分析niche-LR,这是一种将niche-DE统计数据与Niche-net配体-靶矩阵相结合的方法,以识别细胞类型之间的配体-受体信号通道。分析表明Niche-LR可以恢复基于SPOT的空间转录组数据的细胞-细胞信号相互作用,其中每个SPOT可能是细胞的混合物。
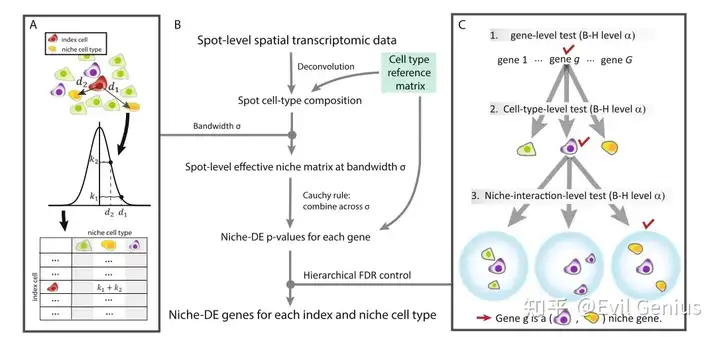
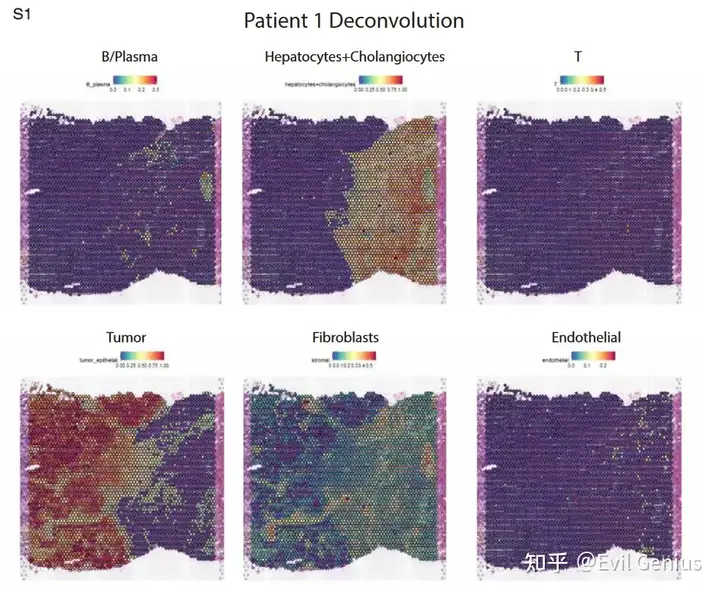
来看看结肠癌的数据分析情况,平台是10X Visium
首先第一步:细胞解卷积,拿到空间细胞类型分布
第二步,识别核心细胞类型类型的Niche-DE:由于成纤维细胞占所有样本细胞的很大一部分,并且由于癌症相关成纤维细胞是抗癌治疗的新兴靶点,因此首先关注成纤维细胞和肿瘤细胞之间的生态位相互作用。成纤维-肿瘤生态位基因的通路富集分析发现,细胞外基质组织、胶原蛋白产生和WNT信号传导是最重要的三个过程,并且研究发现小生境Niche-DE表达的基因确实在成纤维细胞和肿瘤细胞共定位的组织区域富集。

第三步:Niche-LR进行了配体受体分析,在成纤维细胞和肿瘤之间确认的配体受体中检测到了来自肿瘤细胞的25个独特的配体。这包括层粘连蛋白家族基因LAMB1和LAMC1,它们可以与成纤维细胞上的整合素家族受体结合。整合素信号传导已被证明可以控制成纤维细胞的激活和对TME机械信号的反应。
第四步、CODEX 验证
Niche-DE analysis at the single-cell level in CODEX detects a similar pattern of under expression of GAL3 in tumor cells with increasing effective niche presence of fibroblasts。

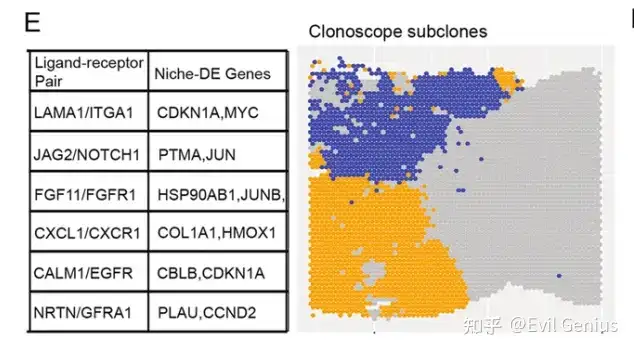
Niche - DE鉴定肿瘤细胞和成纤维细胞之间的亚克隆特异性生态位相互作用
肿瘤细胞表现出很大程度的异质性,这是由于其微环境中内在基因组和表观基因组不稳定性以及外在因素驱动的亚克隆进化。因此,原位鉴定肿瘤细胞的不同亚克隆,并比较这些亚克隆与其局部生态位相互作用的方式,可能使我们了解肿瘤生长和侵袭的机制。这两个亚克隆在亚克隆2中通过染色体4p和9q扩增来区分。
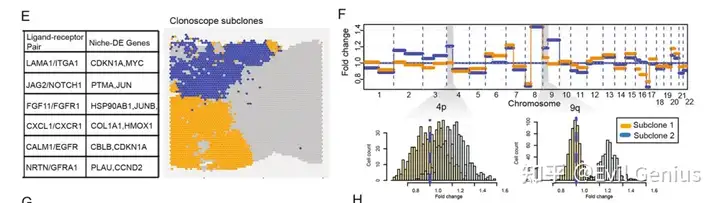
肝样本的组织病理学显示为常规的腺形成腺癌。与含有肿瘤亚克隆1的区域相比,含有亚克隆2的区域具有较少的结缔组织间质,更多的结构复杂性和一些髓样特征,如固体生长模式的病灶。
肿瘤亚克隆2相对于肿瘤亚克隆1的分化程度较低,因为肿瘤亚克隆2的腺体形成更加不规则和减少。将Niche-DE应用于该样本,将成纤维细胞作为索引细胞类型,将两个亚克隆中的每一个作为生态位细胞类型,检测到每个亚克隆特异性的生态位差异表达。亚克隆1附近成纤维细胞标记基因的反应组分析显示干扰素和细胞因子信号的富集。应用以亚克隆1为目标、成纤维细胞为生态位细胞类型的Niche-DE鉴定IFNGR1为亚克隆1(subclone1, fibroblast)+的Niche基因。对亚克隆2附近成纤维细胞标记基因的反应组分析显示糖基化富集和general translation 富集过程。在如此近的空间距离内浸润不同亚克隆的成纤维细胞之间的这些显著差异表明,癌症相关成纤维细胞在适应局部肿瘤微环境的细微变化方面具有高度可塑性.
这里还有其他的示例
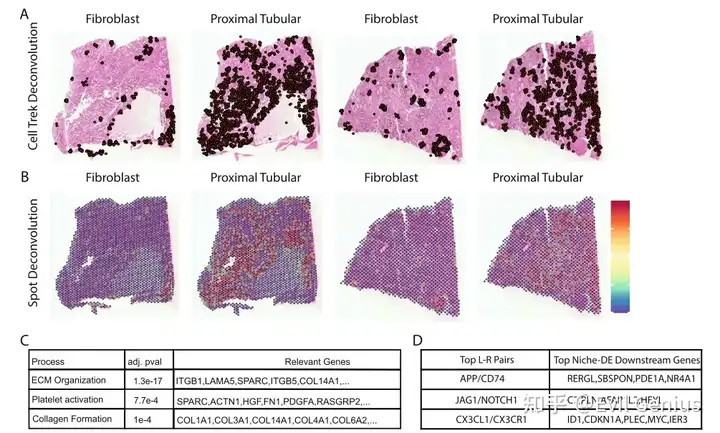
对于SPOT水平的空间转录组数据,在执行Niche-DE之前,一个重要的预处理步骤是反卷积,以估计每个SPOT内的细胞类型比例。Niche-DE检测细胞类型特异性niche基因的准确性依赖于能够区分这些细胞类型的准确性。反卷积不需要匹配的单细胞数据,因为在大多数情况下,同一组织的单细胞参考图谱可用于执行反卷积。
在检测特定细胞类型对之间的生态位基因的Niche-DE敏感性的一个中心因素是其共现率。如果一种细胞类型在空间样本中很少见,那么由于样本量不足,Niche-DE不太可能发现任何信号。如果两种细胞类型从不共定位或总是共定位,则Niche-DE也会分析不足。分析需要观察两种细胞类型在一起和分开的情况,后者为差异表达提供对比。对于SPOT级数据,建议的一个简单规则是,至少有30个SPOT包含生态位细胞类型,并且在其生态位中具有索引细胞类型。
目前,两种最常见的空间转录组数据分析是空间可变基因分析和细胞型共定位分析。从Niche-DE分析得出的结论不包括空间可变基因分析或细胞-细胞相互作用分析。如果一个基因是(i, n) +生态位基因,它的index和生态位细胞类型没有按照一定的空间格局共定位,那么它可能不是一个空间可变基因。此外,索引细胞和生态位细胞不需要随机地共定位,以便在它们之间具有Niche-DE基因。
最后看一眼示例代码
安装
Sys.setenv(R_REMOTES_NO_ERRORS_FROM_WARNINGS="true")
install.packages("devtools")
options(timeout=9999999)
devtools::install_github("Kmason23/NicheDE") # install
library(nicheDE)数据格式
rds转Niche-DE
#read in seurat object
data('liver_met_seurat_object')
#download average expression profile matrix
data("vignette_library_matrix")
data("vignette_deconv_mat")
#make niche-DE object
NDE_obj = CreateNicheDEObjectFromSeurat(liver_met_seurat_object,'Spatial',
vignette_library_matrix,vignette_deconv_mat,sigma = c(1,100,250))矩阵转Niche-DE
#load counts matrix
data("vignette_counts")
#load coordinate matrix
data("vignette_coord")
#load expression profile matrix
data("vignette_library_matrix")
#load deconvolution matrix
data("vignette_deconv_mat")
#make Niche-DE object
NDE_obj = CreateNicheDEObject(vignette_counts,vignette_coord,
vignette_library_matrix,vignette_deconv_mat,
sigma = c(1,100,250))计算和划分Niche(Calculation of the Effective Niche)

NDE_obj = CalculateEffectiveNiche(NDE_obj)Large Scale Data
NDE_obj = CalculateEffectiveNicheLargeScale(NDE_obj,batch_size = 1000, cutoff = 0.05)Niche-DE分析
#Perform Niche-DE
NDE_obj = niche_DE(NDE_obj,num_cores = 4,outfile = "",C = 150, M = 10, gamma = 0.8,print = T, Int = T, batch = T,self_EN = F,G = 1)获取niche-DE的基因
DE_genes = get_niche_DE_genes(NDE_obj,'I',index='stromal',niche = 'tumor_epithelial',positive = T,alpha = 0.05)
head(DE_genes)niche-DE基因的通路富集

#Load enichr package
library(enrichR)
#get fibroblast tumor niche genes
fibro_tum_pos = get_niche_DE_genes(NDE_obj,'I',index='stromal',niche = 'tumor_epithelial',positive = T,alpha = 0.05)
#run pathway enrichment analysis
fibro_tum_processes = enrichr(fibro_tum_pos[,1],databases = 'Reactome_2016')
#View processes in a table
View(fibro_tum_processes$Reactome_2016)Marker Genes

#get marker genes
fibro_tum_markers = niche_DE_markers(NDE_obj,index = 'stromal',niche1='tumor_epithelial',niche2='B_plasma',0.05)
#preview output
head(fibro_tum_markers)最后,配受体分析
data("niche_net_ligand_target_matrix")
data("ramilowski_ligand_receptor_list")
fibro_tumor_LR = niche_LR_spot(NDE_obj,ligand_cell = 'tumor_epithelial',receptor_cell = 'stromal',
ligand_target_matrix = niche_net_ligand_target_matrix,
lr_mat = ramilowski_ligand_receptor_list,K = 25,M = 50,alpha = 0.05,truncation_value = 3)
#preview output
head(fibro_tumor_LR)The output should resemble a 3 column table of ligands,their corresponding receptors, and a list of the top 5 downstream niche-DE genes that drive the ligand potential scoring.
代码示例在https://kmason23.github.io/NicheDE/,供大家研究
NicheDE
生活很好,有你更好
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。