J. Chem. Inf. Model. | ADMET-PrInt药物特性分析平台
J. Chem. Inf. Model. | ADMET-PrInt药物特性分析平台

今天为大家介绍的是来自Sabina Podlewska团队的一篇论文。在新药物的探索过程中,计算策略的发展改变了寻找新药的方式。虽然计算机辅助设计的策略依旧主要集中于确保候选化合物对特定目标的有效性,但是化合物的理化性质及ADMET(吸收、分布、代谢、排泄和毒性)特性的表征已成为计算机辅助药物设计不可或缺的一部分。在这项研究中,作者开发了一款在线应用程序ADMET-PrInt,用于对选定化合物的以下特性进行计算机辅助评估:心脏毒性、溶解度、遗传毒性、膜透过性和血浆蛋白结合能力。除了预测特定属性外,ADMET-PrInt还能够通过解释性方法识别影响这些属性的化合物特征。这对于药物化学家来说至关重要,因为它极大地简化了根据评估属性对化合物结构进行优化的过程。

在药物开发过程中,计算方法的应用极其重要。它们在减少寻找新的生物活性化合物所需的成本和时间上起着关键作用。计算策略主要关注两个方面:评估潜在化合物对特定目标的活性和化合物的理化及ADMET(吸收、分布、代谢、排泄和毒性)特性的评估。迄今为止,已经开发了众多基于机器学习的计算方法来评估化学化合物的ADMET特性。它们通常涉及使用分子描述符和/或指纹捕捉化合物结构和属性,然后使用不同的算法进行分类和回归任务的分析。通过预测不同属性的模型收集,形成了几个ADMET预测包,使得能够生成分析化合物的全面ADMET档案,例如ADMET Predictor、DEREK、MetaPrint2D、MetaPred、SwissADME等等。化学化合物的ADMET表征不仅限于基于配体的方法,还有许多利用基于结构的方法的例子。这些方法涉及到预测化合物与ADMET相关蛋白的相互作用,如细胞色素P450的亚型、hERG钾通道、P糖蛋白、α-1酸性糖蛋白和人血清白蛋白等。在这项研究中,作者开发了ADMET-PrInt:一个用于全面表征化合物在其理化/ADMET属性方面的在线应用程序。考虑的属性包括心脏毒性、溶解度、遗传毒性、膜透过性和血浆蛋白结合。此外,为了帮助药物化学家优化化合物结构,作者开发了解释性方法,旨在促进和指导这一过程。所有工具都在admet.if-pan.krakow.pl上公开提供;作者还分享了代码(在https://github.com/JamEwe/ADMET-PrInt公开提供),该代码可用于开发任何化合物特征的类似预测工具。
方法
数据准备
模拟ADMET属性的数据集是从ChEMBL数据库版本31获取的。为了参考特定的模拟属性,使用了以下实验:
- 膜透过性:Caco2透过性,以μcm/s表示,
- 溶解度:实验ID为CHEMBL3301364,
- 血浆蛋白结合:相应数据以百分比表示,
- 遗传毒性:实验ID为CHEMBL1738317,
- 心脏毒性:以IC50形式展示的hERG通道抑制。
研究中使用的所有数据都是基于人的实验(人肝微粒体、人细胞系等)。此外,为了测试准备好的模型对训练集化学空间之外的化合物的预测能力,形成了一个附加的测试集,该测试集由最新的ChEMBL版本(33)新添加的记录组成,并确定了与训练集中最相关示例的相似性(以Tanimoto系数为准)。为了进行机器学习实验,化学化合物通过图表示和三种类型的指纹表示:由RDKit计算的MACCS、由PaDELPy计算的PubChem和Klekota-Roth(KRFP)。为了加快评估速度并减少噪声信息,对于给定数据集,移除了方差低于固定阈值0.1的指纹位。结果,最终指纹表示的长度根据每个数据集的原始长度而变化,分别为:MACCSFP:166,PubChemFP:472?505,KRFP:647?703。
机器学习模型
在这项研究中作者使用了三种类型的机器学习回归模型来预测ADMET属性。根据先前的研究,采用了以下浅层模型(除非另有说明,否则使用scikit-learn库):岭回归(RR)、随机森林回归器(RF)、基于直方图的梯度提升回归树(HistGrad)、支持向量回归(SVR)、极限梯度提升(XGBoost)和轻量级梯度提升机(LGBM)。除了RR外,所有提到的模型都被用来构建集成模型。最后,对于深度学习模型作者使用了全连接神经网络(FCNN;结合指纹)和图卷积神经网络(GCNN;使用表示化合物的分子图)。超参数选择是通过使用Optuna工具进行的。
评估方法
为了评估每个模型的性能,研究使用了均方误差(MSE)、均方根误差(RMSE)、平均绝对误差(MAE)和决定系数(R^2)指标,并采用了5折交叉验证(CV)的方法。这意味着,数据集中80%的记录构成了训练集,剩余的20%形成了测试集,这个过程重复了5次。
可解释性方法
在预测ADMET属性的同时,ADMET-PrInt还专注于解释这些预测。为此,使用了两种广泛应用的解释性方法:局部可解释LIME)和来自Exmol库的反事实解释(CF)。这两种方法都是模型不依赖的,意味着它们可以用来解释任何提供了输入数据预测结果函数的机器学习模型。尽管这两个包仅提供局部解释(解释给定实例的预测),但在这项研究中实现并使用了各种方法来汇总这些解释,以产生全局解释(解释整个模型)。
结果与讨论
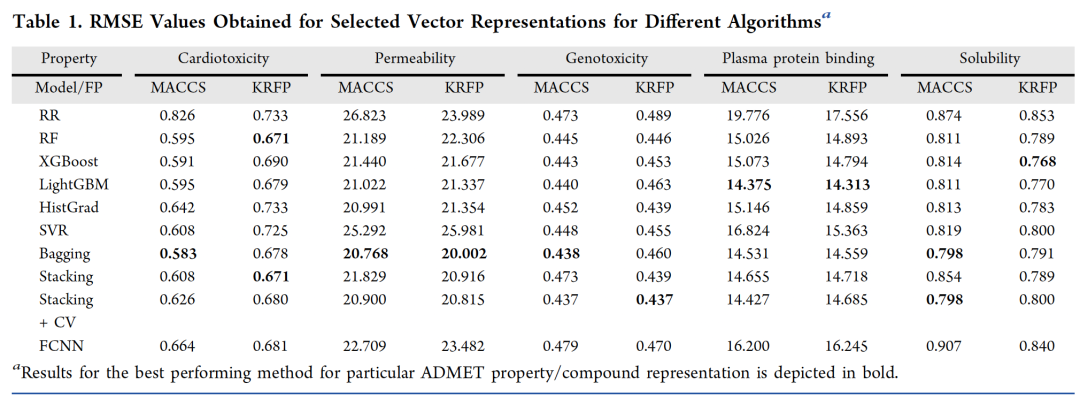
表 1

表 2
在表1中,展示了每种方法对于两种指纹(KRFP和MACCSFP)和分子图所获得的完整RMSE值。基于图和基于指纹实验的总体比较放在了表2中(在每种情况下,都报告了性能最佳的模型)。总体来说,使用图表示和图模型比使用向量表示效果更好,其RMSE值始终较低。然而,在考虑向量表示时,集成模型通常提供了最低的RMSE。详细检查发现,LightGBM在评估血浆蛋白结合率方面(对于MACCS和KRFP)具有最高的预测能力,而RF和XBoost分别对潜在的化合物心脏毒性(对于KRFP)和化合物溶解度(同样对于KRFP)预测最准确。

图 1
作者提供了一种通过示例来展示如何通过修改化合物的特定部分来影响其ADMET属性预测的方法。在这里描述了使用图卷积神经网络(GCNN)模型为选定化合物提供的解释。假设有一种化合物1-(4-chloro-2-(4-chlorophenoxy)phenyl)-N-methylmethanamine,图1显示了为了提高给定ADMET属性预测而建议添加或移除的化学结构。原始化合物的膜透过性预测值为4.304纳米/秒。然而,当一个氯原子被碘原子替代时,预测的透过性下降到了3.628纳米/秒。对氯到氟的替换观察到更大的透过性预测值下降(从4.303降到1.404纳米/秒)。将N-甲基胺变为N-溴胺被预测会导致透过性显著增加至28.252纳米/秒。预测的hERG通道logIC50(心脏毒性)的下降主要通过改变氯苯基部分获得,而通过修改N-甲基胺部分则获得相反的效果。氯苯基和N-甲基胺部分的改变也导致预测的溶解度、遗传毒性和血浆蛋白结合值的变化。这种解释方法有助于研究者理解如何通过修改分子结构来改善或改变其ADMET特性,这对于药物的研发和优化过程至关重要。通过了解哪些修改会提高或降低特定的ADMET属性,研究者可以更有目的地设计新的化合物或优化现有的化合物。
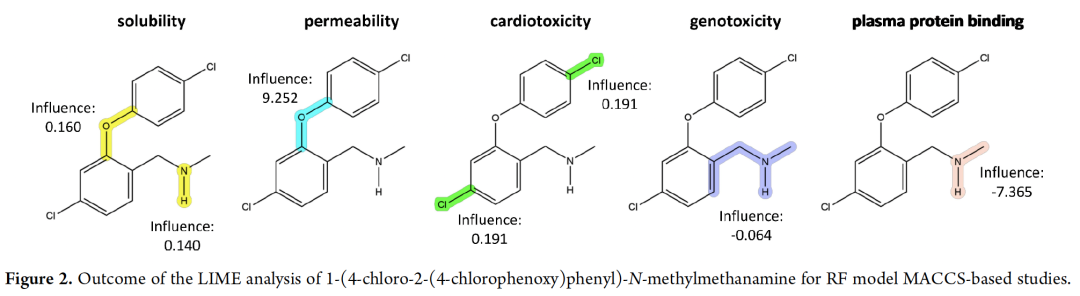
图 2
另一方面,基于LIME的特征分析指出了指纹中相应键编码的功能团,这些功能团对给出特定预测非常重要。图2展示了使用随机森林(RF)模型基于MACCSFP进行的LIME基分析结果。LIME产生的亚结构键对于不同考虑的属性是多样的。在溶解度评估中,包含氮或氧原子的化合物片段被指出为最重要的。对于透过性,含氮环以及两个非碳和非氢原子的连接是得分最高的亚结构。在心脏毒性评估中,氯原子以及含氮和含氧的化合物部分被指出;而对于遗传毒性和血浆蛋白结合,含氮的亚结构和碳原子间有双键的基团被指出。通过这种解释性分析,可以揭示出影响特定ADMET属性预测的关键化学结构,为优化化合物提供了有价值的洞见。这种方法有助于研究人员识别和理解哪些特定的分子部分对药物属性的预测贡献最大,进而可以指导对化合物结构的修改,以改进其理化性质和ADMET特性。
编译 | 曾全晨
审稿 |王建民
参考资料
Jamrozik, E., S?mieja, M., & Podlewska, S. (2024). ADMET-PrInt: Evaluation of ADMET Properties: Prediction and Interpretation. Journal of Chemical Information and Modeling.
代码
https://github.com/JamEwe/ADMET-PrInt