J. Chem. Inf. Model. | 人工智能增强多物种肝脏微粒体稳定性预测
J. Chem. Inf. Model. | 人工智能增强多物种肝脏微粒体稳定性预测

今天给大家介绍一篇由中南大学湘雅药学院曹东升教授团队在Journal of Chemical Information and Modeling近期发表的关于肝微粒体稳定性性质预测模型的文章《Enhancing Multi-species Liver Microsomal Stability Prediction through Artificial Intelligence》。该文献通过整合多个数据库的数据,构建了一个庞大的多物种肝微粒体稳定性数据集,并利用机器学习算法构建了106个共识模型。通过SHAP方法和原子热图分析,揭示了影响肝微粒体稳定性的重要特征。研究还应用MMPA方法和亚结构衍生算法,提取了与肝微粒体稳定性相关的分子转化规则。这项研究为药物研发领域提供了新预测模型和分子解释,为药物设计和筛选提供了重要的指导和支持。

引言
在当今药物研发领域,准确评估药物在不同物种中的肝微粒体稳定性对于药物代谢和毒性评估至关重要。肝微粒体是肝细胞内质网膜上的小囊泡,承载着大部分药物代谢过程中的关键酶系统,如细胞色素P450酶。了解药物在肝微粒体中的稳定性可以帮助预测药物的代谢速率和生物利用度,从而指导药物研发的方向和策略。然而,传统的实验方法耗时耗力,且成本高昂,限制了大规模药物筛选和评估的效率。因此,利用计算机辅助预测模型成为一种重要的解决方案。过去的研究表明,基于机器学习和人工智能的肝微粒体稳定性预测模型在药物研发中具有潜在的应用前景。这些模型可以快速准确地评估大量化合物的代谢稳定性,为药物设计和筛选提供重要参考。然而,现有的肝微粒体稳定性预测模型往往受限于单一物种或特定数据类型的依赖,同时缺乏跨物种比较和实际的模型解释。
因此,该研究旨在构建适用于人类、大鼠和小鼠的多物种肝微粒体稳定性预测模型,并提供全面的解释和分析。通过整合多个代表性数据库的数据,构建庞大且可公开访问的数据集,结合不同的分子表征方式和机器学习算法,研究者们致力于提高预测模型的性能和泛化能力,希望为药物研发领域提供更准确、高效的肝微粒体稳定性评估工具,推动新药的发现和开发。工作的整体工作流程如图1所示。
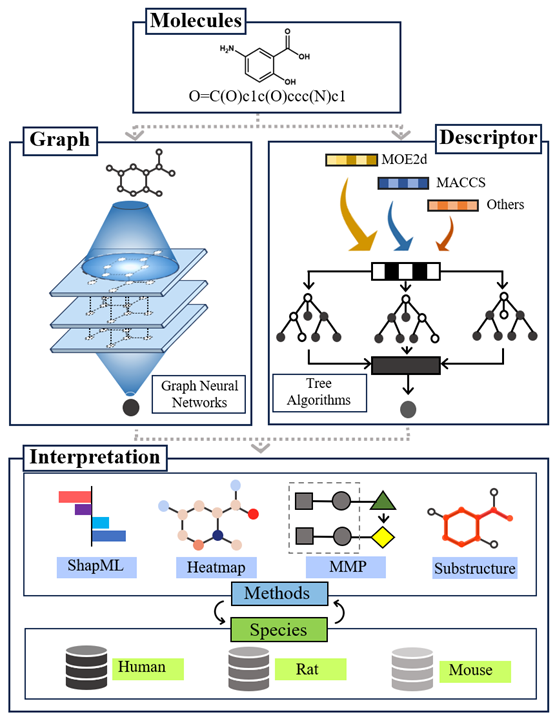
图1. 工作流程
材料与方法
首先,研究者通过广泛搜索代谢相关分子信息,从多个数据源中收集了肝微粒体稳定性的多物种数据,包括人类、大鼠和小鼠。随后,对这些数据进行多步骤的预处理,包括去除无结构信息的化合物、标准化处理、消除重复标签等。最后,创建了一个包括不同物种数据的庞大数据集,包括15,344个人类数据、9,601个大鼠数据和1,978个小鼠数据。
在分子表征方面,研究者使用了分子图以及多种描述符,包括MOE2d、CATS、MACCS等描述符的计算。在模型构建与超参数优化阶段,他们使用了8种机器学习算法构建肝微粒体稳定性预测模型,包括传统的随机森林、XGBoost、支持向量机和梯度提升树等算法,以及基于图的深度学习算法如图卷积网络、图注意力网络等。针对不同算法,采用了不同的超参数优化方法,如蛮力方法和Parzen估计器树(TPE)方法,以提高模型性能。最后,通过验证集评估训练模型的性能,并进行外部测试集的比较,以验证模型的准确性和适用性。
结果与讨论
模型结果
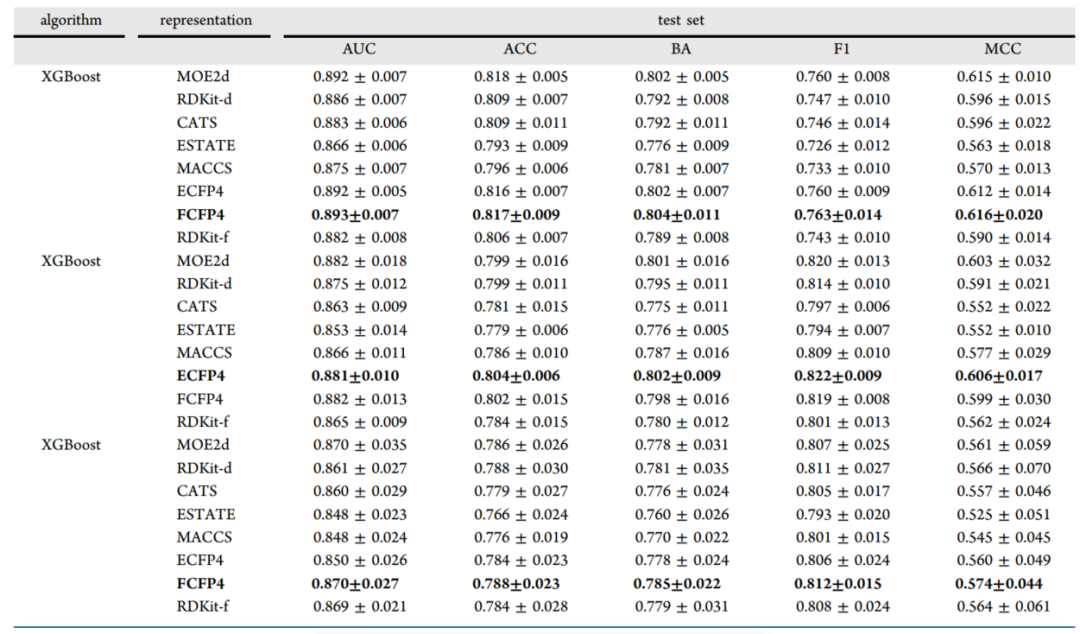
表1. 肝微粒体稳定性模型在人类、大鼠和小鼠物种测试集上的表现
研究者基于不同的分子表征方式和机器学习算法,在3个物种上建立了一共108个肝微粒体稳定性分类模型。模型结果如表1所示,与基于图的模型相比,基于描述符的模型具有更高的预测性能。在HLM模型中,结合XGBoost算法和FCFP4指纹的模型表现出了最好的性能,测试集的AUC值为0.893,MCC值为0.616。RLM模型中, XGBoost算法和MOE2d描述符的组合性能最佳,在测试集上的MCC值为0.603。同时,结合XGBoost算法和FCFP4指纹的MLM模型性能最佳,在测试集上的MCC值为0.574。
通过结合已有的算法和描述符,研究者们还构建了106个共识模型。共识模型的结果表明,共识模型始终优于基于单个描述符集或单个算法构建的模型。当使用相同的算法并适当组合不同的描述符时,肝微粒体稳定性共识模型的预测能力可以进一步提高。
与其他过滤规则和模型的比较

表2. 多物种肝微粒体稳定性模型与其他平台肝微粒体稳定性模型的性能比较
研究者们利用额外收集的多物种肝微粒体数据作为公平比较的外部测试集。如表2所示,研究者们将建立的最佳共识模型与现有的肝微粒体稳定性预测模型(如ADME@ NCATS和vNN-ADMET)进行了比较。结果表明,研究者构建的肝微粒体稳定性模型在各自物种数据集上预测性能最好,对人类、大鼠和小鼠的预测ACC值分别为0.757、0.804和0.763。
模型解释
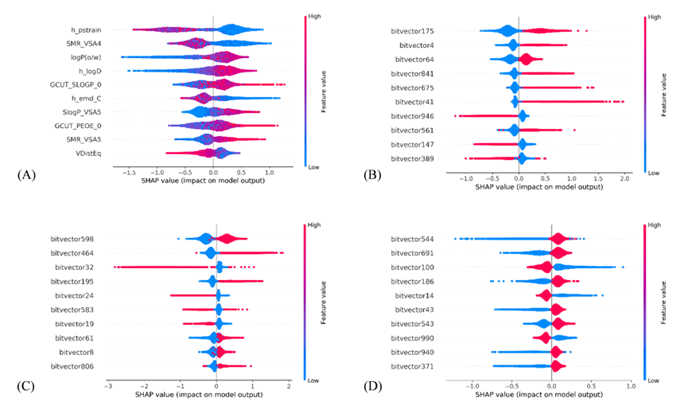
图2. HLM模型中最重要的10个分子描述符及其对应的SHAP值:(A) MOE2d描述符。(B) ECFP4指纹图谱。(C) FCFP4指纹图谱。(D) RDKit指纹图谱。
研究者选择了SHAP方法对不同物种构建的部分模型进行了整体的解释分析,结果如图2所示。SHAP方法基于Shapley值理论,通过量化每个特征对整体预测的贡献,可以深入了解每个样本的预测结果。除了解释每个物种对应的肝微粒体稳定性模型外,研究者还比较了多物种模型,用来表现物种间差异性对构建模型的影响。

图3. 基于Attentive FP模型解释机制的原子热图和影响HLM条目中 (A)假阳性化合物和(B)假阴性化合物的结果输出的重要分子描述符分布图
为了更好地理解预测整体模型与具体预测之间的差异,研究者还选择了一些有代表性的分子,使用分子在模型输出提供的描述符SHAP值和Attentive FP算法的可解释层提供的原子热图进行展示,结果如图3所示。结果表明,如果分子没有正确地学习先前的模型分析范式或者范式规则本身适用范围较小,那么在预测过程中可能会导致该分子被错误预测。
数据解释
研究者利用匹配分子对(MMPs)方法和先前开发的子结构生成算法,进一步揭示了分子亚结构转化与肝微粒体稳定性之间的关系,部分结果如表3、表4所示。研究者进行分析发现,一些显著的转化规律包括了单原子转化和多原子片段转化,子结构算法提取的代表性肝微粒体稳定性亚结构有效补充了与先前模型解释不同的结构知识。不同的物种具有不同的代表性转化规则和亚结构,强调了构建肝微粒体的稳定性模型需要重视实验数据来源的物种差异性。
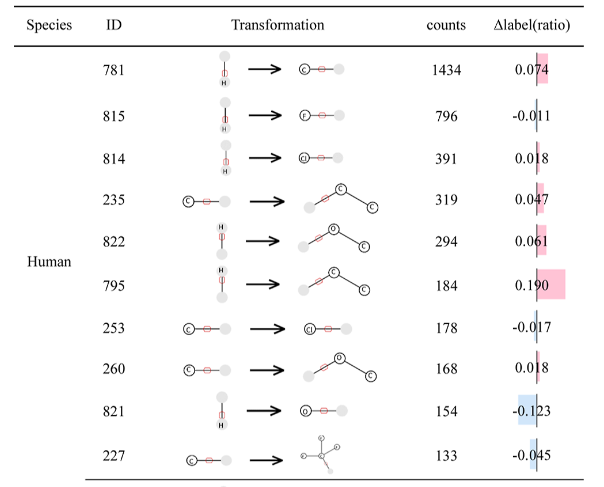
表3. 影响人类肝微粒体分子稳定性的代表性匹配分子对化学转化
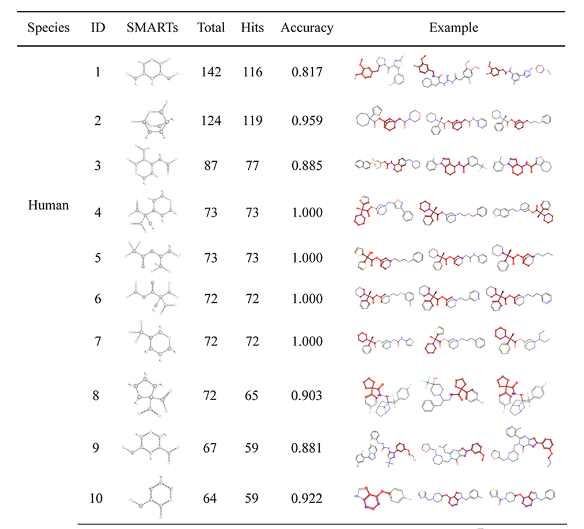
表4. 影响人类肝微粒体分子稳定性的代表性亚结构
总结
研究团队指出,通过构建多种肝微粒体稳定性预测模型,包括基于描述符和基于图的模型,能够有效预测人类、大鼠和小鼠三种不同物种的肝微粒体稳定性。基于图的深度学习算法在预测精度和泛化能力上表现优异,尤其是在不同物种中的预测效果较好。通过组合不同描述符和机器学习算法构建共识模型,进一步提高了模型的预测性能和泛化能力。同时,SHAP等解释方法在模型中的应用,为理解不同物种肝微粒体稳定性的差异性提供了新的视角,有助于优化药物设计和筛选过程,推动新药的发现和开发。
参考资料
Long, Teng-Zhi, De-Jun Jiang, Shao-Hua Shi, You-Chao Deng, Wen-Xuan Wang, and Dong-Sheng Cao. "Enhancing Multi-species Liver Microsomal Stability Prediction through Artificial Intelligence." Journal of Chemical Information and Modeling (2024).