生信技能树Day9 GEO数据挖掘 差异分析
原创生信技能树Day9 GEO数据挖掘 差异分析
原创用户11064093
发布于 2024-04-21 11:42:42
发布于 2024-04-21 11:42:42
差异分析表格
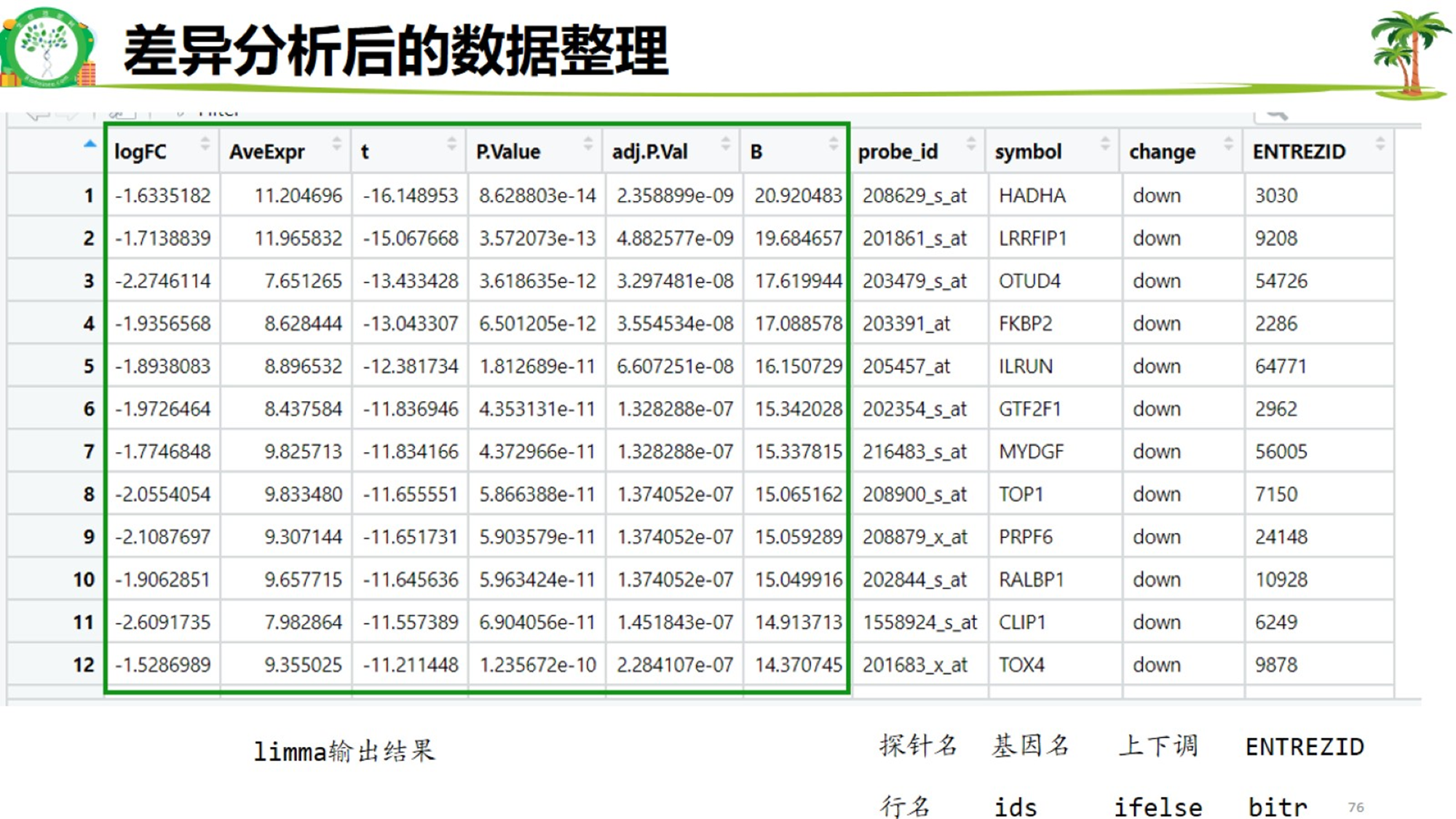
差异分析后要得到这样一张表格
二分组数据差异分析
#差异分析 limma
library(limma)
design = model.matrix(~Group) # 生成模型矩阵
fit = lmFit(exp,design)
fit = eBayes(fit)
deg = topTable(fit,coef = 2,number = Inf)分组多代码更复杂
为deg数据框添加几列
1.加probe_id列,把行名变成一列
library(dplyr)
deg = mutate(deg,probe_id = rownames(deg))2.加上探针注释
因为探针和基因注释不是一对一的关系,所以要去重

# 随机去重
ids = distinct(ids,symbol,.keep_all = T)
deg = inner_join(deg,ids,by="probe_id")
nrow(deg) #如果行数为0就是你找的探针注释是错的。#保留最大值
exp2 = exp[ids$probe_id,]
identical(ids$probe_id,rownames(exp2))
library(dplyr)
ids = ids %>%
mutate(exprowsum = rowSums(exp2)) %>%
arrange(desc(exprowsum)) %>%
select(-3) %>%
distinct(symbol,.keep_all = T)
nrow(ids)
# 拿这个ids去inner_join#求平均值
exp3 = exp[ids$probe_id,]
rownames(exp3) = ids$symbol
exp3[1:4,1:4]
exp4 = limma::avereps(exp3)
# 此时拿到的exp4已经是一个基因为行名的表达矩阵,直接差异分析,不再需要inner_join 3.加change列,标记上下调基因
logFC_t = 1
p_t = 0.05
#思考,如何使用padj而非p值
k1 = (deg$P.Value < p_t)&(deg$logFC < -logFC_t)
k2 = (deg$P.Value < p_t)&(deg$logFC > logFC_t)
deg = mutate(deg,change = ifelse(k1,"down",ifelse(k2,"up","stable")))
table(deg$change)火山图-----------------------------------------------------------------
library(ggplot2)
ggplot(data = deg, aes(x = logFC, y = -log10(P.Value))) +
geom\_point(alpha=0.4, size=3.5, aes(color=change)) +
scale\_color\_manual(values=c("blue", "grey","red"))+
geom\_vline(xintercept=c(-logFC\_t,logFC\_t),lty=4,col="black",linewidth=0.8) +
geom\_hline(yintercept = -log10(p\_t),lty=4,col="black",linewidth=0.8) +
theme\_bw()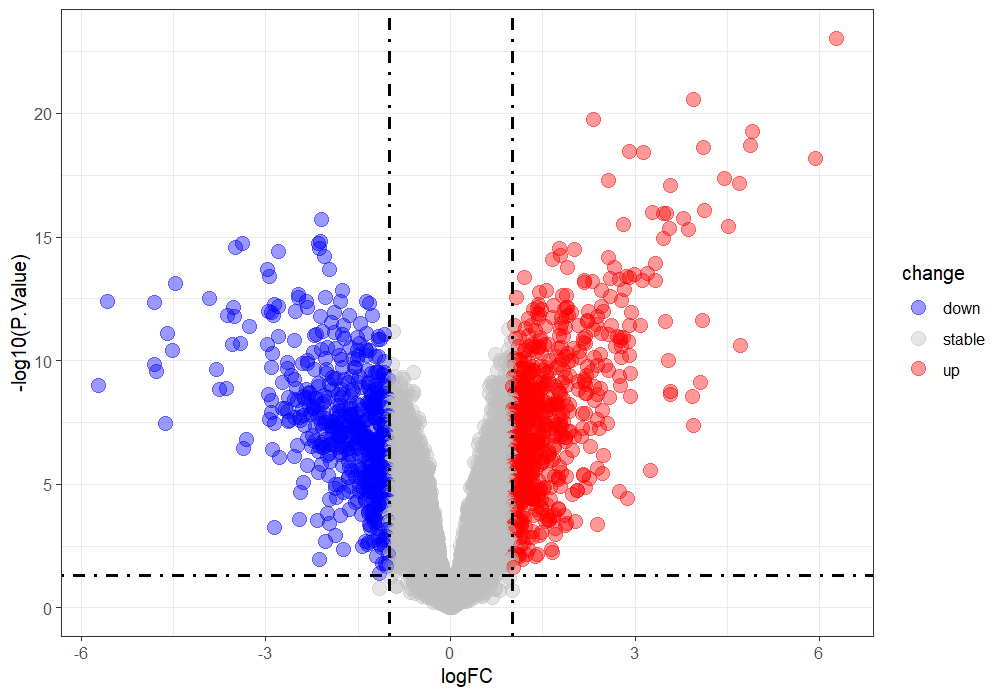
火山图
差异基因热图------------------------------------------------------------
# 表达矩阵行名替换为基因名
exp = exp[deg$probe_id,]
rownames(exp) = deg$symbol
diff_gene = deg$symbol[deg$change !="stable"]
n = exp[diff_gene,]
library(pheatmap)
annotation_col = data.frame(group = Group)
rownames(annotation_col) = colnames(n)
pheatmap(n,show_colnames =F,
show_rownames = F,
scale = "row",
#cluster_cols = F,
annotation_col=annotation_col,
breaks = seq(-3,3,length.out = 100)
) 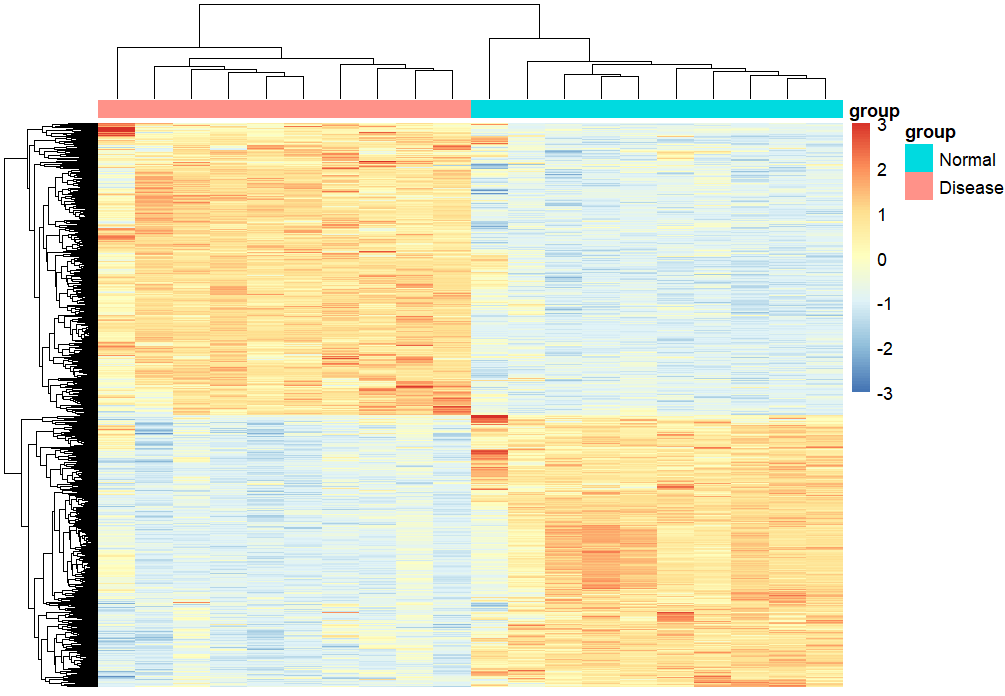
差异基因热图
重要的是学会画图思路,先找到示例代码,根据自己的数据和需求修改,好好阅读帮助文档。先把示例数据跑通,再把自己的数据改成示例数据的格式,最后修改参数。
4.加ENTREZID列,用于富集分析(symbol转entrezid,然后inner_join)
library(clusterProfiler)
library(org.Hs.eg.db)
s2e = bitr(deg$symbol,
fromType = "SYMBOL",
toType = "ENTREZID",
OrgDb = org.Hs.eg.db)#人类,注意物种
#一部分基因没匹配上是正常的。<30%的失败都没事。
#其他物种http://bioconductor.org/packages/release/BiocViews.html#___OrgDb
nrow(deg)
deg = inner_join(deg,s2e,by=c("symbol"="SYMBOL"))
#多了几行少了几行都正常,SYMBOL与ENTREZID不是一对一的。
nrow(deg)
save(exp,Group,deg,logFC_t,p_t,file = "step4output.Rdata")富集分析
# 准备工作
rm(list = ls())
load(file = 'step4output.Rdata')
library(clusterProfiler)
library(ggthemes)
library(org.Hs.eg.db)
library(dplyr)
library(ggplot2)
library(stringr)
library(enrichplot)(1)输入数据
gene_diff = deg$ENTREZID[deg$change != "stable"] (2)富集
ekk <- enrichKEGG(gene = gene_diff,organism = 'hsa')
ekk <- setReadable(ekk,OrgDb = org.Hs.eg.db,keyType = "ENTREZID")
ego <- enrichGO(gene = gene_diff,OrgDb= org.Hs.eg.db,
ont = "ALL",readable = TRUE)
#setReadable和readable = TRUE都是把富集结果表格里的基因名称转为symbol
class(ekk)(3)可视化
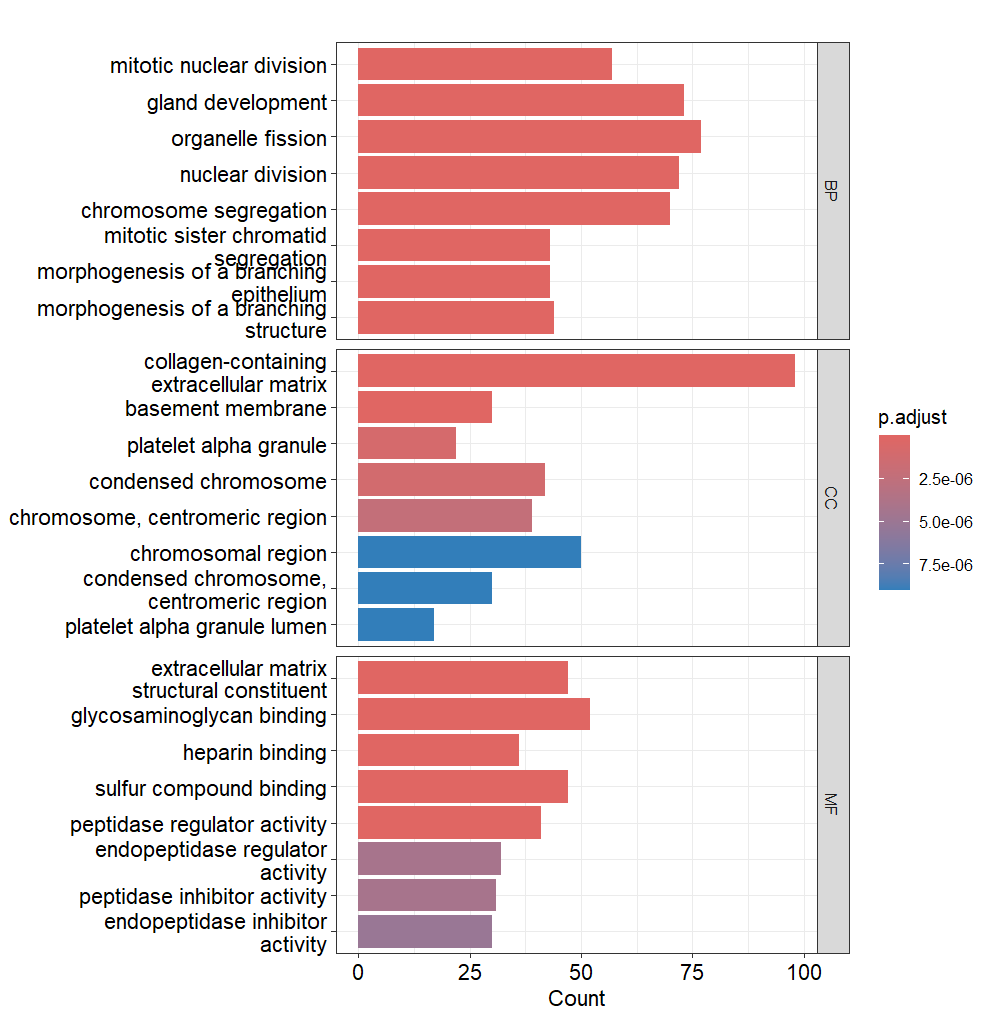
barplot(ego, split = "ONTOLOGY") +
facet_grid(ONTOLOGY ~ ., space = "free_y",scales = "free_y")
barplot(ekk)
# 默认用的是adjustP值,可以自己去对象里找原始P值用ggplot2画图
#或者是dotplot
# 更多资料---
# GSEA:https://www.yuque.com/docs/share/a67a180f-dd2b-4f6f-96c2-68a4b86fe862?#
# Y叔的书:http://yulab-smu.top/clusterProfiler-book/index.html
# GOplot:https://mp.weixin.qq.com/s/LonwdDhDn8iFUfxqSJ2Wew
# 网上的资料和宝藏无穷无尽,学好R语言慢慢发掘~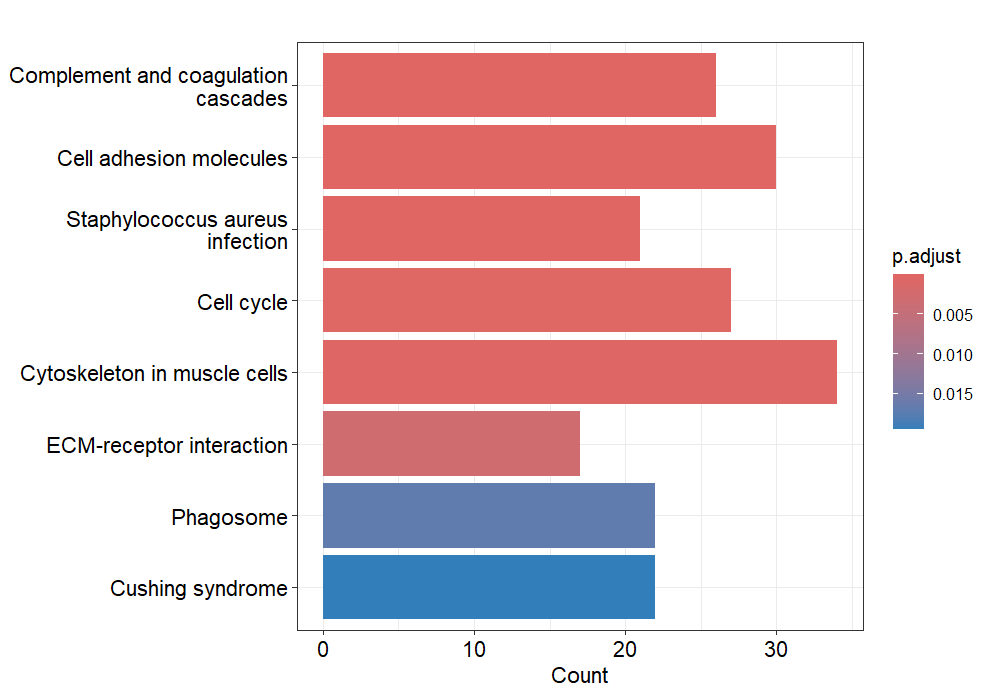

生信技能树
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录