整合多模态空间组学数据开源框架--SpatialData
原创整合多模态空间组学数据开源框架--SpatialData
原创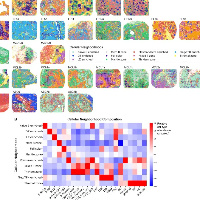
引言
在当今生命科学领域,空间组学技术(spatial omics technologies)已成为揭示生物组织结构与功能复杂交互关系的重要工具。这些技术通过在组织特定位置对DNA、RNA、蛋白质以及代谢物的定量分析,使研究人员能够以前所未有的分辨率和全面性理解生物组织的分子组成和空间结构。然而,伴随空间组学数据量的爆炸式增长以及数据类型的多样化,如何高效地处理、整合以及分析这些大规模的空间组学数据集成为了该领域面临的重要挑战。为应对这一挑战,一种名为SpatialData的开放式和通用数据框架应运而生(3月20日 Nature Methods “SpatialData: an open and universal data framework for spatial omics”)。这一框架旨在为空间组学数据提供一个统一和可扩展的多平台文件格式,同时提供对超出内存大小的数据延迟加载、数据转换和对常用坐标系统的对齐等功能。通过SpatialData,研究人员可以方便地进行空间注释、跨模态聚合分析,极大地提升了空间组学数据的可用性和分析效率。空间组学结合了成像和分子分析技术,可以在细胞乃至亚细胞水平上定位和量化分子,揭示细胞在组织中的精确位置及其相互作用。然而,不同的空间组学技术,如基于荧光显微镜的成像技术和基于测序的空间转录组学,往往产生不同格式和类型的数据,这些数据的差异性为数据的集成和综合分析带来了难题。SpatialData框架通过建立一个统一的数据格式和程序接口来解决这一问题,使得来自不同来源和技术的空间组学数据可以被统一处理和分析。此外,该框架还支持对数据进行延迟加载和多尺度展示,这对于处理大规模数据集尤为重要。通过SpatialData,研究人员可以轻松地在多个数据模态之间进行对齐和集成分析,推动对生物系统空间组织结构的深入理解。

生物组织的功能与其构成和组织密切相关。成像和空间分子分析技术的进步使得通过对组织结构进行全面、高分辨率和高灵敏度的探究成为可能。现有的空间分子分析方法能够实现原位定量DNA、RNA、蛋白质和/或代谢物的丰度。其中一些技术采用光学显微镜,提供了从亚细胞到整个生物体的长度尺度的形态特征的空间分辨率。空间组学技术发展迅速,各种数据模态和方法具有不同的优势和局限性,例如在空间分辨率、分子多样性和检测灵敏度范围等方面的权衡。有效整合和操作来自不同空间组学模态的数据对于构建生物系统的整体视图至关重要。
尽管在分析单个空间组学数据集方面已经取得了进展,但统一和多模空间组学数据的整合涉及到一些重要的实际挑战,这些挑战并未得到现有解决方案的充分解决。即使是基本操作,如将数据集以一致的方式加载到分析管道中,也会受到数据类型的多样性(例如,用于测序的表格数据和用于图像的数十到数百千兆字节的密集数组)和文件格式(例如,特定技术供应商格式)的阻碍。
此外,单个空间组学模式在空间分辨率和组织数据采集的空间区域方面可能存在巨大差异,并且通常不会对齐。因此,为了整合这些数据,它们必须被适当地转换和对齐到一个共同的坐标系统(CCS),这是建立全球通用坐标框架(CCF)的前提条件。最后,解开多模空间组学数据集的复杂性需要专业知识和动力,以实现大规模交互式数据探索和注释的方法。因此,为了释放新兴空间多组学研究的全部潜力,需要计算基础设施来存储、探索、分析和注释跨越空间组学技术的全部范围的数据,并提供统一的程序接口。
1、SpatialData的设计概览和核心功能
统一文件格式(Unified File Format):为了克服不同空间组学平台和数据类型的多样性,SpatialData引入了一个统一和可扩展的文件格式,这个格式允许数据的互操作性(interoperability)和重用(reusability)。这种格式建立在开放显微镜环境-下一代文件格式(OME-NGFF)规范上,并利用Zarr文件格式进行存储,从而支持传统文件系统和云存储(cloud-based storage)的高效访问。空间元素(Spatial Elements)和坐标系统(Common Coordinate Systems):SpatialData通过定义五种基本的空间元素(Spatial Elements)——图像(Images)、标签(Labels)、点(Points)、形状(Shapes)和表格(Tables)——来表示空间数据集。此外,它通过跟踪应用于单个数据集的坐标转换或对齐步骤,支持不同数据集的有效对齐和转换到共同坐标系统。延迟加载(Lazy Loading)和数据操作:SpatialData对象在内存中表示这种格式,支持大于内存数据的延迟加载,这对于处理大型数据集尤其重要。Python库还提供了用于数据访问、对齐、查询和聚合的操作,这些操作允许用户定义生物组织的CCS,并在不同的模态间部署空间查询和聚合操作。交互式注释和数据可视化:SpatialData集成了一个napari插件,用于交互式数据注释和可视化。用户可以利用这个插件定义空间注释,如绘制感兴趣区域(ROIs)或为多数据集注释指导地标。此外,SpatialData还提供了生成交云以及静态图的功能,以促进数据的理解和表达。深度学习集成:通过实现PyTorch数据集类,SpatialData使得直接从空间组学数据对象训练深度学习模型成为可能,这为使用空间组学数据进行机器学习和深度学习提供了基础。

2、乳腺癌三个空间数据集的对齐和综合分析(示例)
为了说明SpatialData在多模态集成和分析中的实用性,分析使用该框架来表示和处理一项乳腺癌研究的数据,该研究结合了苏木精和伊红(H&E)图像以及10x Genomics Visium和Xenium检测。该研究包括两个原位测序数据集(Xenium)和一个空间转录组学数据集(10x Visium CytAssist),这些数据集来自乳腺癌肿瘤连续切片。首先,分析使用napari-spatialdata来定义所有数据集中存在的地标点,然后使用变换对所有三个数据集进行对齐以定义一个CCS。通过对齐的结果,SpatialData使分析能够识别出共同的空间区域,可以通过跨数据集的SpatialData查询访问。
接下来,分析利用来自所有三个数据集的集体信息创建了一组共享的空间注释。简要地说,根据H&E图像中的组织学特征,使用napari-spatialdata选择了四个感兴趣区域(ROI)。然后,利用Visium中的全基因组转录组信息估计拷贝数状态(使用CopyKat18)并注释主要的遗传亚克隆。最后,通过从独立的乳腺癌单细胞RNA测序(scRNA-seq)图中转移细胞类型标签(在scanpy14中实现)来注释两个Xenium复制品中的细胞类型。
为了说明SpatialData如何用于在数据集之间转移空间注释,分析考虑了Visium捕获位置的掩码,并从重叠的Xenium细胞中聚合细胞类型信息,以估计每个位置的细胞类型比例。为了比较,还考虑了基于细胞2位置20的Visium计数的去卷积分析,使用了与同一来源的scRNA-seq细胞类型19作为参考。
分析观察到Xenium复制品之间细胞类型丰度估计的高一致性(在Visium位置上的中位数Pearson相关系数为0.88),以及Xenium和基于去卷积的估计之间的整体良好一致性(中位数Pearson相关系数为0.69)。与在Visium位置进行聚合类似,分析考虑了从H&E定义的ROI和从Visium的子克隆位置联合定义的区域。再次,在每个区域内量化了细胞类型的分数,可以直接使用来自Xenium的细胞计数分数,也可以通过解卷积对应的Visium数据来实现。两个Xenium复制品显示出细胞类型分数的高一致性,而且Xenium和Visium也是一致的。
作为第二个聚合使用案例,分析比较了在Visium捕获位置上使用Xenium或Visium数据对个别基因的表达估计。分析再次将Visium捕获位置转移到聚合Xenium中的个别分子计数到Visium掩模中。如预期的那样,聚合计数在Xenium复制品之间高度一致,在Xenium和Visium计数之间也是如此。分析还注意到整体转录丰度与不同组织切片和技术之间的一致性之间存在直接关系。

癌症三个空间数据集的比对和综合分析
开发团队使用napari-spatialdata定义所有数据集中存在的地标点,然后使用变换对齐所有三个数据集来定义通用坐标系(CCS)。对齐的结果显示:SpatialData 能够识别公共空间区域,可以使用跨数据集的SpatialData 查询来访问该空间区域。接下来,利用来自所有三个数据集的集体信息创建了一套共享的空间注释。最后,举例说明如何使用SpatialData在数据集之间传输空间注释和聚合功能,这些示例说明了聚合功能的灵活性,可以在不同类型的spatialelement(点、圆形捕获位置、细胞和更大的解剖ROIs)之间应用,以传输不同类型的空间注释(细胞表达、细胞类型分数)。
总之,SpatialData框架提供的灵活性和随时可用的解决方案为分析提供了新的可能,并提高了综合空间分析的可重复性。
随着SpatialData使用率的不断提高,其实用性也将进一步增强,正在进行的开发扩展了SpatialData与R/Bioconductor的互操作性,提供对多尺度点和多边形表示的支持,并支持以编程方式和通过可视化工具Vitessce进行基于云的数据访问。
SpatialData是通过pip作为Python包提供的,并附带了大量的示例和教程,可从如下文档中访问:
https://spatialdata.scverse.org.



生活很好,有你更好
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。