榕树集--AlphaFold 3: 解锁生命密码!预测各类分子结构与互作!
榕树集--AlphaFold 3: 解锁生命密码!预测各类分子结构与互作!


简介
人类是一个精密的工具,其内部由无数的分子机器,不断的运转,提供能量,修饰,翻译,等等作用。这些机器主要由蛋白质,核酸,以及其余分子等等组成。如何预测,观察他们的相互作用,才能够为我们改造世界,提供精准的参考意见。
时光飞逝,日月如梭,Alphafold 1和2的引入,推动了蛋白质的结构以及蛋白--蛋白相互作用建模的革命。而最近的RFAA验证了,在一个统一的深度学习框架下,实现蛋白质--小分子,蛋白质--核酸等等,生物分子复合物建模的可能性。之前也描述过一个AF-latest的过渡的版本,当时是以新闻的方式推出,没有代码,同样也可以进行不同生物分子的复杂建模。
所以,今天的重点就是AF3,其基于新的扩散的架构,并且在准确性上优于许多先前的工具,也就是AF3实现了在统一的深度学习框架下,进行不同生命分子的高精度建模。
具体的一些参考阅读链接放在文后。
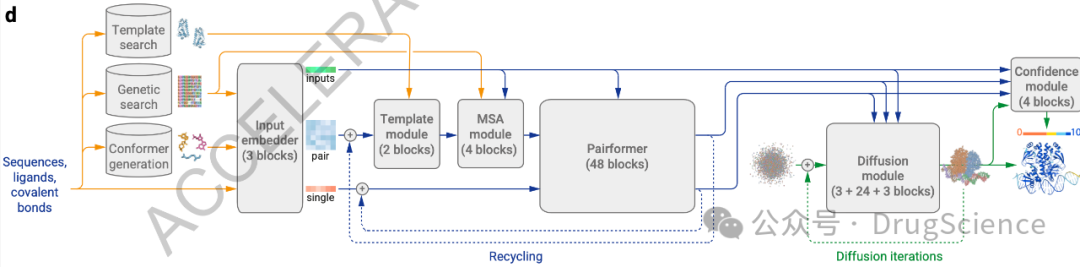
框架改进和训练
AF3相比于专门针对特定任务的强大方法,在蛋白质结构和蛋白质相互作用结构的准确性方面取得了显著提高。这一成就得益于对AlphaFold 2架构和训练程序的实质性演变,以适应更广泛的化学结构,并提高了学习的数据效率。AF3采用了类似于AlphaFold 2的整体架构,但在各个主要组件中都有较大的差异。对于处理多重序列比对(MSA)的方式进行了重大改进,采用了更小、更简单的MSA嵌入块。AF3还引入了新的Pairformer模块,取代了AlphaFold 2中的Evoformer模块,用于处理成对表示和单一表示,而不保留MSA表示。最终,AF3使用新的扩散模块直接操作原子的原始坐标,而不是AlphaFold 2中的结构模块。这种扩散模块的操作方式有助于提高预测的准确性,并通过生成式训练过程产生答案的分布,从而避免了对残基进行基于扭转的参数化以及结构上的违例损失。
使用生成式扩散方法所面临的最大的问题是生成模型可能会在非结构化区域产生虚假结构,即幻觉。为了解决这个问题,作者采用了一种新颖的交叉蒸馏方法,在训练数据中添加了AlphaFold-Multimer v2.3预测的结构,以减少幻觉的发生。另外,作者还开发了置信度指标,用于预测最终结构中的原子级和成对误差,并介绍了用于训练这些指标的方法。最后,作者对模型进行微调以提高性能的方法。
预测精度
AlphaFold 3(AF3)在各种生物复合物结构预测准确率:
- 蛋白质-配体界面:在PoseBusters基准测试集上,AF3的准确率为口袋与配体的根均方偏差小于2 ?。据报道,AF3在此基准测试集上的准确率高于传统对接工具如Vina和Gold。
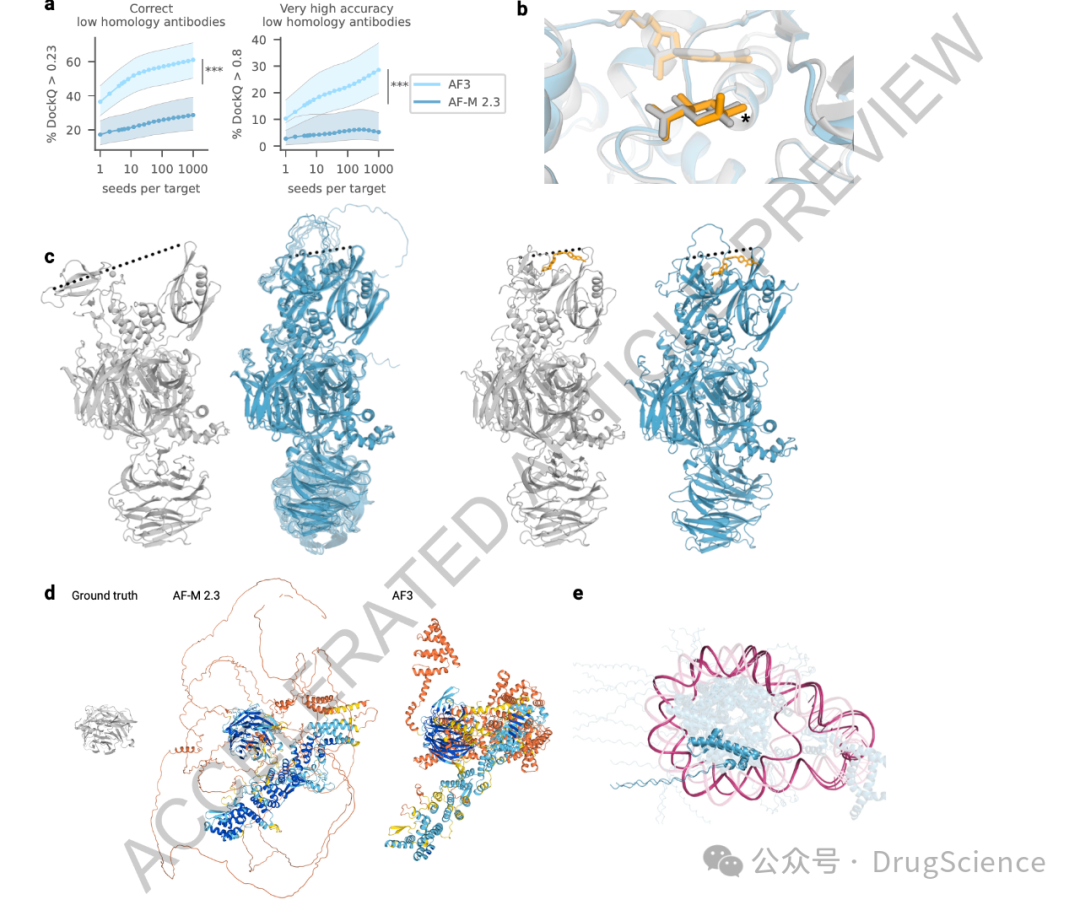
- 蛋白质-核酸复合物和RNA结构:AF3在预测蛋白质-核酸复合物和RNA结构时,相较于RoseTTAFold2NA模型,具有更高的准确率。具体数字可能需要参考下图。
- 共价修饰:AF3能够准确预测共价修饰,包括键合配体、糖基化和修改的聚合物残基。准确率可能因不同类型的共价修饰而有所不同。
- 蛋白质复合物准确性:AF3相较于AlphaFold-Multimer v2.3(AF-M 2.3),具有更高的蛋白质复合物准确性。
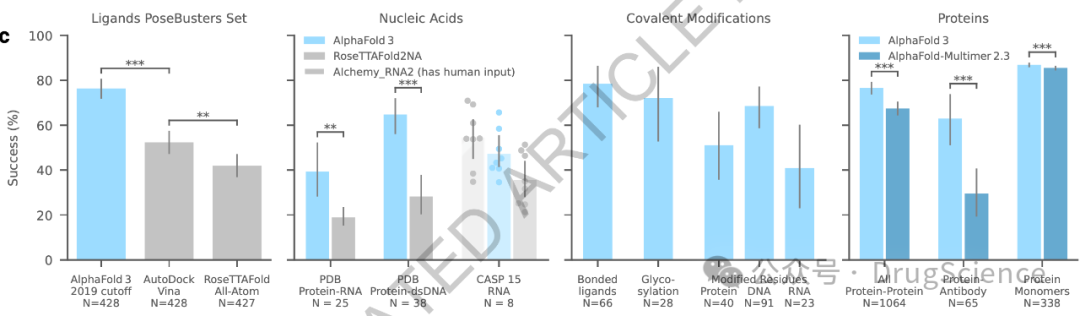
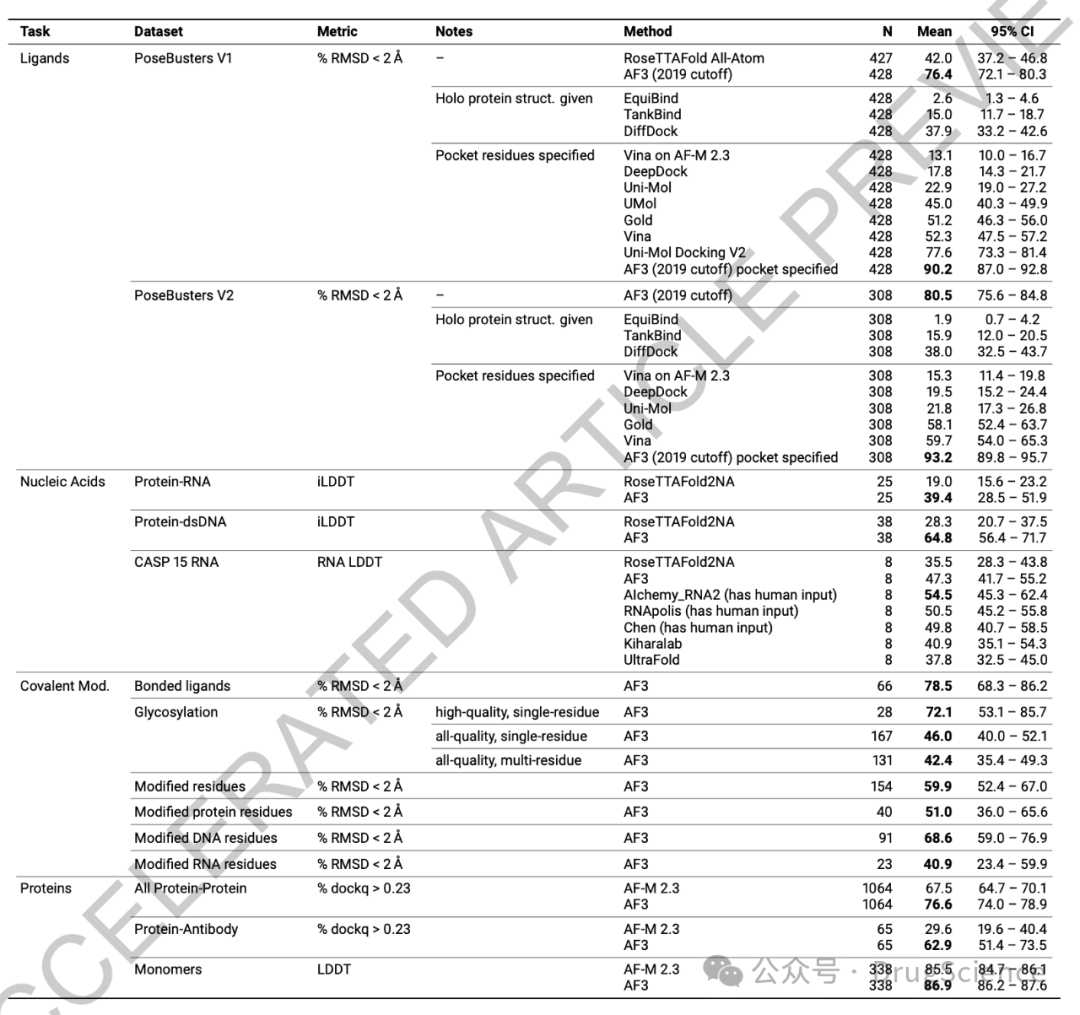
对于配体和核酸,N表示结构的数量;对于共价修饰和蛋白质,N表示cluster的数量。
模型限制
模型在立体化学、幻觉、动态和特定目标的准确性方面仍然存在一些限制。在立体化学方面,模型存在手性违规和原子重叠(冲突)的问题,尽管已经采取了一些措施来减轻这些问题。其次,由于从AlphaFold 2模型转换到基于扩散的AlphaFold 3模型,模型容易在无序区域产生虚假结构(幻觉),但已经采取了措施来解决这个问题。此外,模型无法准确预测生物分子系统在溶液中的动态行为,并且在某些情况下,模型建模的构象状态可能不正确或不全面。最后,尽管AlphaFold 3模型在建模准确性方面取得了巨大进步,但仍然存在一些目标的准确建模可能具有挑战性,可能需要生成大量的预测并对其进行排名。
Code:no,Server:yes
目前来说,Deepmind团队推出的AF3是世界上预测分子间相互作用的最精准的工具。谷歌提供了一个平台,而并没有更新其github上的仓库,其希望科学家使用其平台服务来进行生命分子的结构建模(proteins, DNA, RNA, and a selection of ligands, ions, and chemical modifications)。
服务地址:https://www.alphafoldserver.com/
估计这几天人多
总结
AlphaFold 3模型在预测生物分子机器上取得了重大进展,展示了在统一框架内准确预测各种生物分子系统的结构是可能的。虽然预测所有类型相互作用的准确性仍然存在挑战,但该模型展示了强大的覆盖能力和泛化能力。同时,它强调了开发合适的深度学习框架可以大幅减少获得生物学相关性能所需的数据量,并放大已收集数据的影响。因为实验结构测定方法的持续进步,例如冷冻电镜和层析成像的显著改进,将提供大量新的训练数据,进一步提高这些模型的泛化能力。
参考
Abramson, J., Adler, J., Dunger, J. et al. Accurate structure prediction of biomolecular interactions with AlphaFold?3. Nature (2024). https://doi.org/10.1038/s41586-024-07487-w