主动学习加速分子模拟中的几何构型优化,附ASE优化器python包下载
主动学习加速分子模拟中的几何构型优化,附ASE优化器python包下载
编译/文龙
机器学习正在重塑许多科学和工程领域的研究方法。在表面催化领域,机器学习的各种应用不断涌现,能够对分子进行更广泛的模拟,包括分离研究、结构优化、力场的动态学习和高通量筛选。
最近,卡内基梅隆大学的研究人员提出了一种基于神经网络集成的主动学习方法,可以同时加速多个分子模拟中的局部几何构型优化,计算数量减少了 50-90%,使研究人员能够在更少的时间内完成相同的工作。
研究结果于 6 月 17 日以「Machine-learning accelerated geometry optimization in molecular simulation」为题发表在期刊《The Journal of Chemical Physics》上。

分子的几何构型优化是计算材料和表面科学的重要组成部分,因为它是寻找基态原子结构和反应的途径,可用于估计分子和晶体结构的热力学和动力学特性。理论上,这一过程是缓慢的,因为它涉及使用 DFT 这类量子化学方法对力进行迭代计算,需要大量的算力和时间。
因此,研究团队通过将先验数据集成到主动学习框架中,实验说明了通过使用神经网络集成在不同配置之间共享信息,与具有单一配置的主动学习相比,具有多种配置的主动学习可以实现进一步的加速,新模型将密度泛函理论(DFT)和有效介质理论(EMT)计算的调用量减少了 50% 至 90%。
此外,该团队提供了一个原子模拟环境 (ASE) 优化器的 Python 包,以便更轻松地使用神经网络集成主动学习进行几何构型优化。
加速大量配置的主动学习的潜在方法
论文的通讯作者 John R. Kitchin 表示:「这项工作的另一个目标是概述基于神经网络的在线学习在各种局部几何构型优化任务上的性能。」
该团队从两个方面加速主动学习过程:一是限制用于在每一步更新智能体模型的训练集的大小;二是不同弛豫(relaxation)轨迹的信息可以相互共享以加速整体弛豫过程。
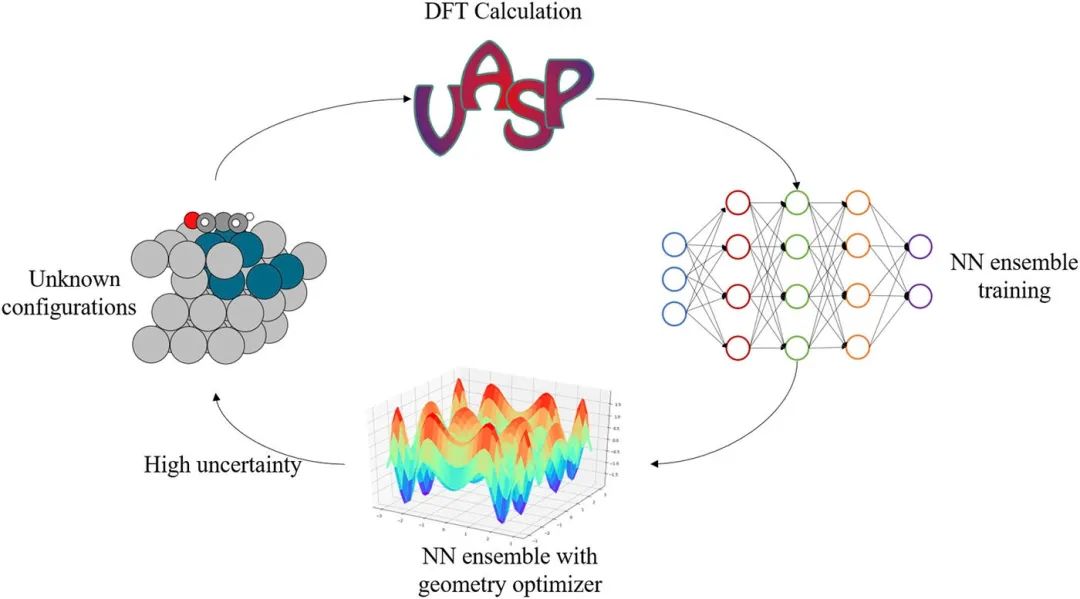
整个工作流程从需要弛豫的初始配置开始。首先,计算 DFT 能量和力,并使用此初始信息训练神经网络。然后,该模型与优化器一起使用以减少配置的能量。当遇到不确定的配置或达到弛豫标准时,模型的弛豫停止。提交不确定的配置以进行进一步的 DFT 计算。
Kitchin 进一步解释说:「通常,当我们进行几何构型优化时,我们会从头开始,使得计算很少从我们过去所知道的事情中受益。通过在过程中添加智能体,我们使其能够依赖先前的计算,而不是每次都从头开始。」
论文链接:http://dx.doi.org/10.1063/5.0049665
项目地址:https://github.com/lmj1029123/SingleNN
参考内容:https://phys.org/news/2021-07-geometry-optimization-molecular-simulation.html
人工智能?×?[?生物 神经科学?数学 物理 材料 ]
「ScienceAI」关注人工智能与其他前沿技术及基础科学的交叉研究与融合发展。
欢迎关注标星,并点击右下角点赞和在看。
点击阅读原文,加入专业从业者社区,以获得更多交流合作机会及服务。