“站长,热图咋画?”——有关pheatmap包的一般用法及代码解读
“站长,热图咋画?”——有关pheatmap包的一般用法及代码解读
热图 就是很热的图,会冒火的那种图~~~数据挖掘文章必备
少废话,直接上代码
软件平台:R(3.4.3)library(pheatmap)library(RColorBrewer)library(ggsci)library(DESeq2)vsd.T <- vst(dds, blind = FALSE) 选取差异基因做热图 resSig_P <- subset(res, abs(log2FoldChange)>1 & padj < 0.01)mat.1 <- assay(vsd.T.1[rownames(resSig_P), ])mat.1 <- mat.1 - rowMeans(mat.1) 选取区分明显的基因做热图 topVarGenes <- head(order(rowVars(assay(vsd.T)), decreasing = TRUE),1000)mat <- assay(vsd.T[ topVarGenes, ])mat <- mat - rowMeans(mat) 设置行列名 anno.1 <- as.data.frame(colData(vsd.T.1)[, c("condition")])rownames(anno.1) <- colnames(mat.1)colnames(anno.1) <- c("State")anno.1_T=subset(anno.1,anno.1
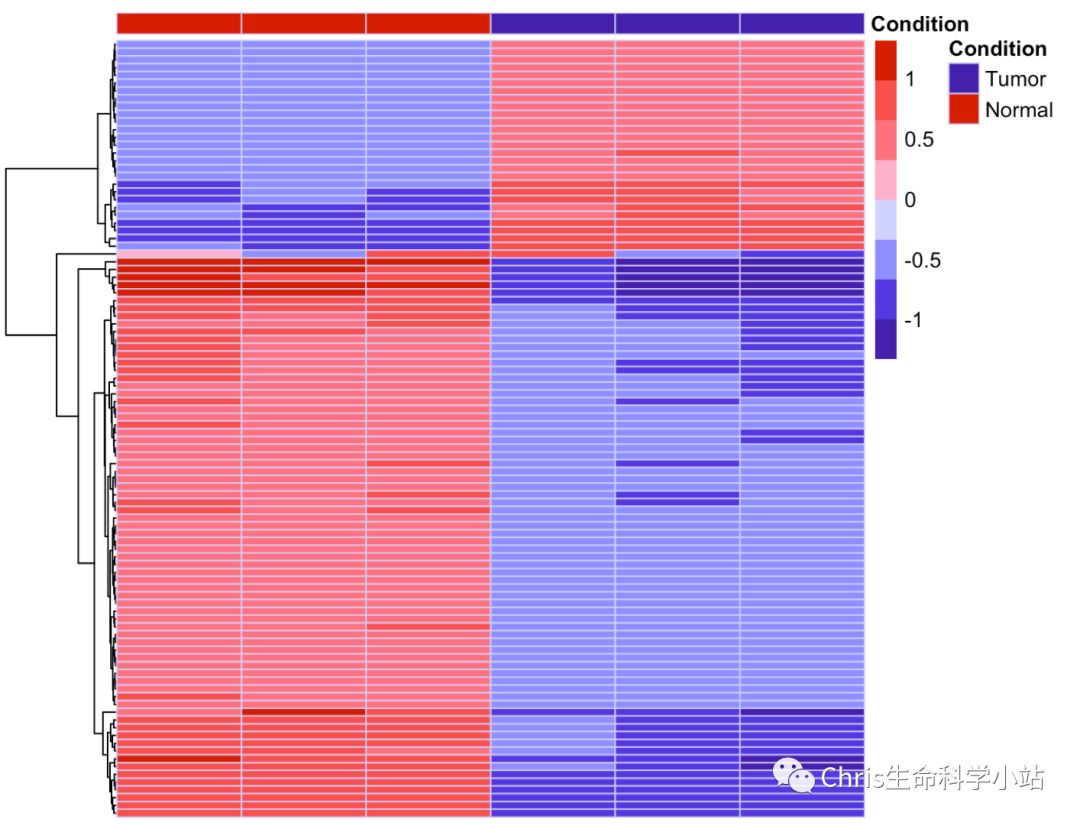
代码解释
1resSig_P <- subset(res, abs(log2FoldChange)>1& padj <0.01)这句中res为DESeq2输出的结果,来自于这个:res=result(dds),另外,倍数和padj参数自己看心情调整吧2topVarGenes <- head(order(rowVars(assay(vsd.T)), decreasing = TRUE),1000)这句选择变化明显的基因,其中1000这个数字自己看心情调整吧3mat <- mat - rowMeans(mat)这句就是热图中数值标准化算法,当然还有别的,想DIY自己去google吧4anno.1 <- as.data.frame(colData(vsd.T)[, c("condition")])这句中"condition"的condition就是DESeq2准备的coldata中那个列名,这个名字错了会错5pheatmap(mat.1, annotation_col = anno.1,cluster_cols = F,cluster_rows =F,show_colnames=F,show_rownames = F, annotation_colors = ann_colors,color = mypal) 这句中所有的F都可以改成T,F就是关闭,T就是打开比如列聚类打开cluster_cols=T,行聚类打开cluster_rows =T显示列名show_colnames=T,显示行名show_rownames = T6rownames(anno.1) <- colnames(mat.1)colnames(anno.1) <- c("State")anno.1_T=subset(anno.1,anno.1State=="Tumor")anno.1_N=subset(anno.1,anno.1