10Xvisium HD高精度平台探索
原创10Xvisium HD高精度平台探索
原创
追风少年i
发布于 2024-04-21 13:40:50
发布于 2024-04-21 13:40:50
作者,Evil Genius
看到最近的反馈,大家似乎对相亲的事情很感兴趣啊, ,但是大家别光顾笑啊,女孩子想想自己30多岁会不会也是这样,男孩子30多了想想遇到这样的女人该怎么办。
尤其女孩子,一定要想好自己的变化,当初本科的时候没有一个女同学说自己看重物质,甚至鄙视那些只看物质的女人,但是等真到了这个时候,就会变成自己当初讨厌的样子,别看91年的大姐现在要车要房直接嫁,甚至被人称作伏弟魔,人家读书的时候也是希望靠奋斗、靠自己的,只不过岁月改变了你我。
也不是说没有好女孩子,我的一个同学就娶了一个我认为是男生理想的对象,长相甜美,身高167,体重100出头,96年的,我同学出了16.8万的彩礼,人家女方说结婚是两个人的事,出那么多彩礼干什么,然后就陪送了26.8万的嫁妆,外加一辆车,要知道我那同学挫男一个,身高169,跟帅完全不沾边,而且跟我一样93年的,两人相亲2个多月就结婚了,前几天说孩子快生了,要我去庆祝一下,说实话,这混蛋让我一直想跳楼, ,但这种女孩子真的可遇不可求。
有对象的,好好珍惜,没有的,努力找一个,趁着身边人还没有被污染的时候, 。
高精度平台带来的最大的最明显的变化,就是微环境分析,包括邻域与社区。还有就是算力变化,为了提升分析能力,需要转移到python中分析了。

目前scanpy也更新了对于HD的分析,我们来简单看一下:
import scanpy as sc
import pandas as pd
from anndata import AnnData
from pathlib import Path
import json
from matplotlib.image import imread
from step import scModel, stModel
from step.utils.misc import read_visium_hd
sc.set_figure_params(dpi=150, figsize=(6, 4.5))Visium HD Human Colorectal Cancer (16 um) cell type clustering & spatial domain identification
16 um bin size: cell type clustering
adata = read_visium_hd("./data/visium-hd/human-coloretal-cancer/square_016um/")
stepc = scModel(
adata=adata,
n_top_genes=2000,
)
stepc.run(epochs=400, batch_size=2048, beta=1e-3)
adata = stepc.adata
sc.pp.neighbors(adata, use_rep='X_rep', n_neighbors=60)
sc.tl.umap(adata)
sc.tl.leiden(adata)
sc.pl.umap(adata, color='leiden')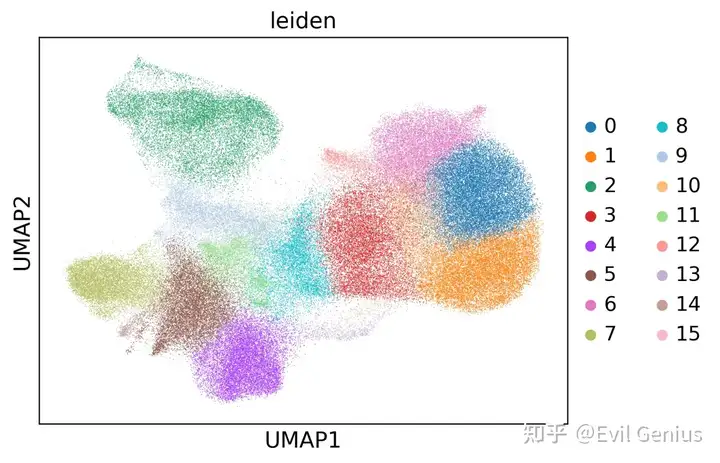
sc.pl.spatial(adata, color='leiden')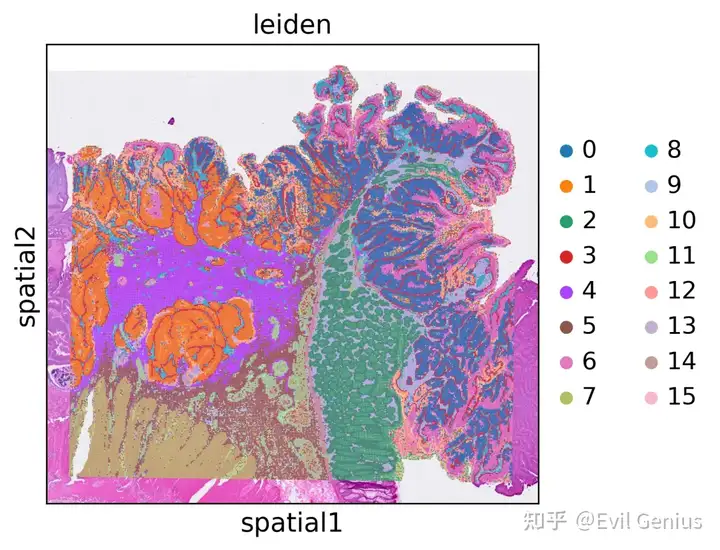
stepc.save("./results/visium-hd/hcc-16um/")
adata.write_h5ad("./results/visium-hd/hcc_16um.h5ad")
adata = sc.read_h5ad("./results/visium-hd/hcc_16um.h5ad")
# sc.set_figure_params(dpi=300, dpi_save=300)
# sc.settings.figdir = './results/visium-hd/hcc-16um/'
sc.pl.spatial(adata, color='leiden', frameon=False, groups=['7', '11', '14', '2', '9', '10'], na_in_legend=False)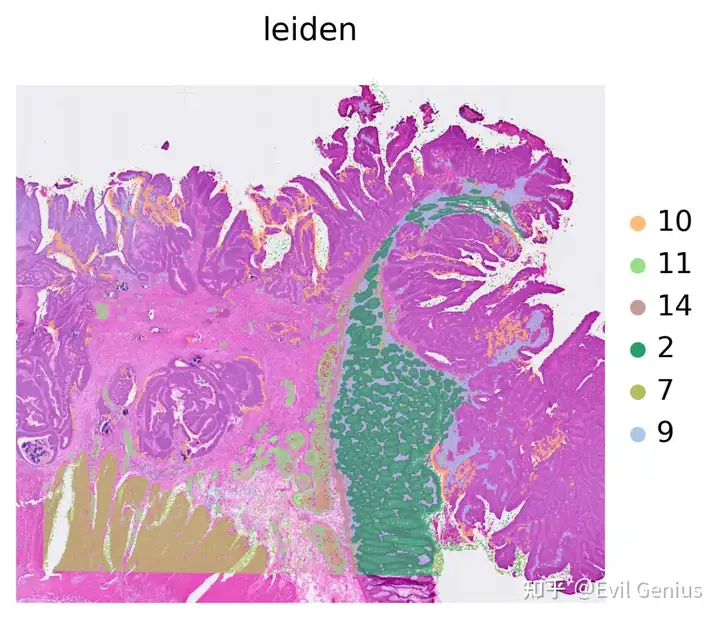
sc.settings.figdir = "./results/visium-hd/hcc-16um/"
sc.set_figure_params(dpi_save=300)
sc.tl.rank_genes_groups(adata, groupby='leiden', dendrogram=False, use_raw=True)
sc.tl.dendrogram(adata, use_raw=True, groupby='leiden')
WARNING: You’re trying to run this on 2000 dimensions of `.X`, if you really want this, set `use_rep='X'`.
Falling back to preprocessing with `sc.pp.pca` and default params.
sc.pl.rank_genes_groups_matrixplot(adata,
groupby='leiden',
values_to_plot='logfoldchanges',
cmap='RdBu_r',
vmin=-4, vmax=4,
save='16um_leiden.svg')
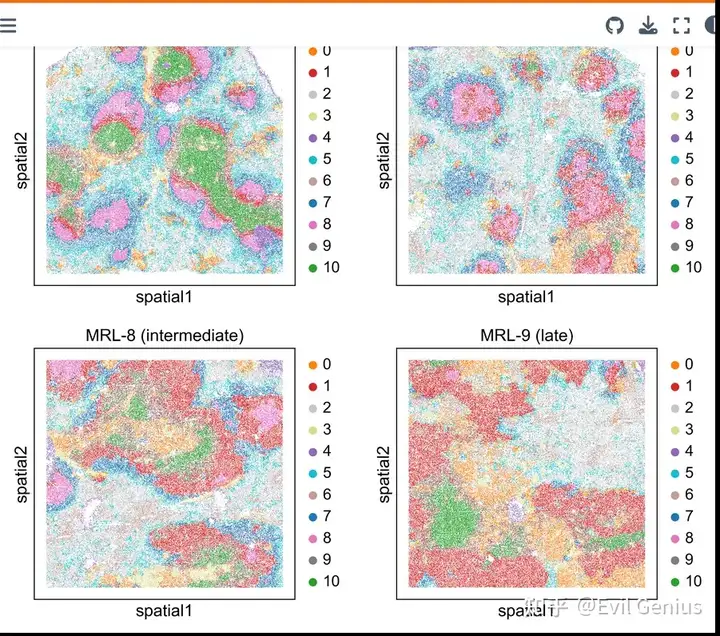
16 um bin size: spatial domain identification
adata = read_visium_hd("./data/visium-hd/human-coloretal-cancer/square_016um/")
stepc = stModel(
adata=adata,
n_top_genes=3000,
edge_clip=2,
n_glayers=3,
)
stepc.cluster(n_clusters=12)
stepc.spatial_plot(color='domain',)
stepc.save("./results/visium-hd/hccspatial_plotomain/")
stepc = stModel.load("./results/visium-hd/hccspatial_plotomain/",
filepath="./results/visium-hd/hcc_16um_domain.h5ad")
stepc.adata.obsm['X_smoothed'] = stepc.gembed()
stepc.cluster(n_clusters=8)
stepc.spatial_plot(color='domain',)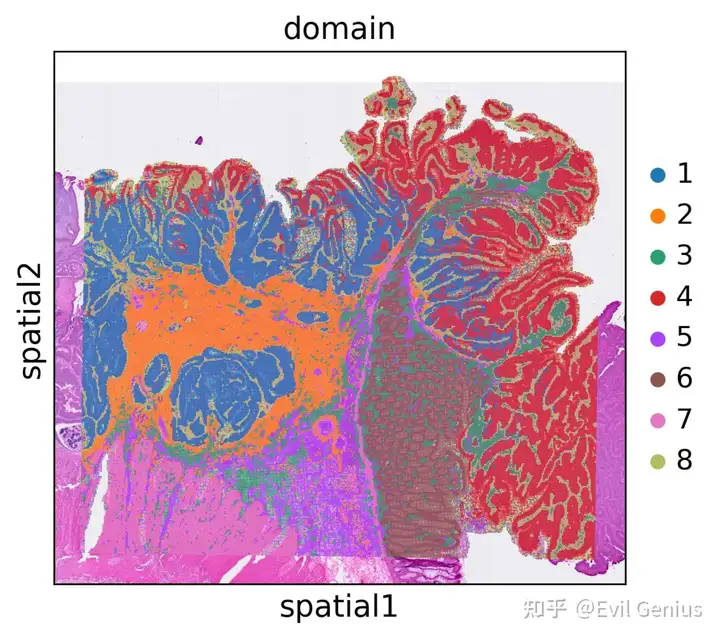
stepc.adata.write_h5ad("./results/visium-hd/hcc_16um_domain.h5ad")
adata = sc.read_h5ad("./results/visium-hd/hcc_16um_domain.h5ad")
sc.settings.figdir = "./results/visium-hd/hcc-16um/"
sc.set_figure_params(dpi_save=300, figsize=(12, 9))
sc.pl.spatial(adata, color='domain', frameon=False, show=False, save='_16um_domain.svg',)
adata_raw = adata.raw.to_adata()
sc.pp.log1p(adata_raw)
adata.raw = adata_raw
# adata = stepc.adata
sc.tl.rank_genes_groups(adata, groupby='domain', dendrogram=False, use_raw=True)
sc.tl.dendrogram(adata, use_raw=True, groupby='domain')
sc.pl.rank_genes_groups_matrixplot(adata,
groupby='domain',
values_to_plot='logfoldchanges',
cmap='RdBu_r',
vmin=-4, vmax=4,
save='16um_domain.svg')
Visium HD Mouse Small Intestine (8 um and 16 um) cell type clustering & spatial domain identification
import scanpy as sc
from step import scModel, stModel
from step.utils.misc import read_visium_hd
sc.set_figure_params(dpi=150, figsize=(6, 4.5), dpi_save=300)16 um bin size: Cell type clustering
adata = read_visium_hd("./data/visium-hd/mouse-intesitine/square_016um/")
stepc = scModel(
adata=adata,
n_top_genes=2000,
)
stepc.run(epochs=400, batch_size=1024,)
adata = stepc.adata
sc.pp.neighbors(adata, use_rep='X_rep', n_neighbors=60)
sc.tl.umap(adata)
sc.tl.leiden(adata)
sc.pl.umap(adata, color='leiden')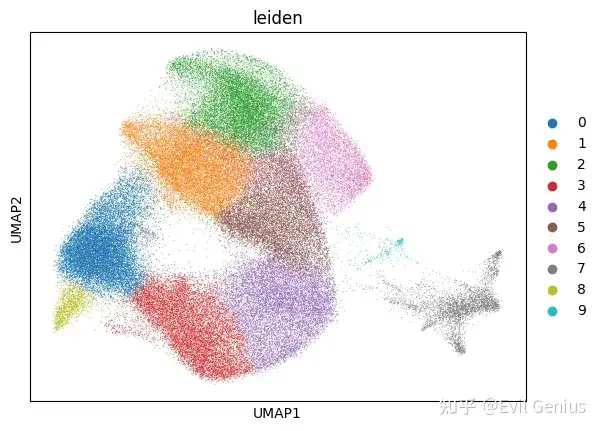
sc.pl.spatial(adata, color='leiden')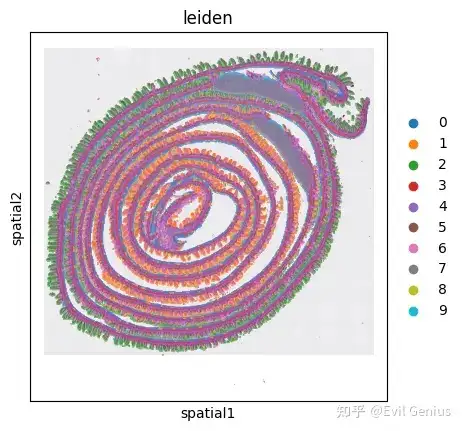
stepc.save("./results/visium-hd/config")
adata.write_h5ad("./results/visium-hd/mouse_intesitine_16um.h5ad")
adata = read_visium_hd("./data/visium-hd/mouse-intesitine/square_008um/")
stepc = stModel(
adata=adata,
n_top_genes=2000,
edge_clip=1,
)
stepc.run()
stepc = stModel.load("./results/visium-hd/mouse-intestine/config-8um/",
filepath='./results/visium-hd/mouse_intesitine_8um.h5ad')
stepc.cluster(n_clusters=8)
stepc.spatial_plot(color='domain')
import torch
torch.cuda.empty_cache()
stepc.add_embed(key_added='X_rep')
adata = stepc.adata
sc.pp.neighbors(adata, use_rep='X_rep', n_neighbors=60)
sc.tl.umap(adata)
stepc.save("./results/visium-hd/mouse-intestine/config-8um")
adata.write_h5ad("./results/visium-hd/mouse_intesitine_8um.h5ad")
adata = sc.read_h5ad("./results/visium-hd/mouse_intesitine_8um.h5ad")
sc.pl.umap(adata, color='domain', show=False, save='8um_domain.svg')
adata = stepc.adata
sc.tl.rank_genes_groups(adata, groupby='domain', method='wilcoxon', dendrogram=False, use_raw=True)
sc.tl.dendrogram(adata, groupby='domain')sc.settings.figdir = "./results/visium-hd/mouse-intestine/config-8um/"
sc.pl.rank_genes_groups_matrixplot(adata,
groupby='domain',
values_to_plot='logfoldchanges',
cmap='RdBu_r',
use_raw=True,
vmin=-4, vmax=4, save="8um_domain.svg")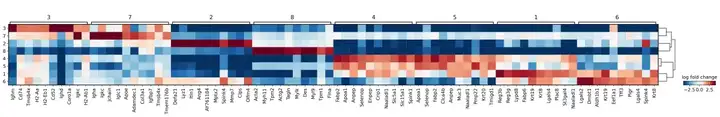
8 um bin size: Cell type clustering
adata = read_visium_hd("./data/visium-hd/mouse-intesitine/square_008um/")
stepc = scModel(
adata=adata,
n_top_genes=2000,
)
stepc.run(epochs=400, batch_size=4096)
adata = stepc.adata
sc.pp.neighbors(adata, use_rep='X_rep', n_neighbors=60)
sc.tl.umap(adata)
sc.tl.leiden(adata, resolution=0.8)
sc.pl.umap(adata, color='leiden')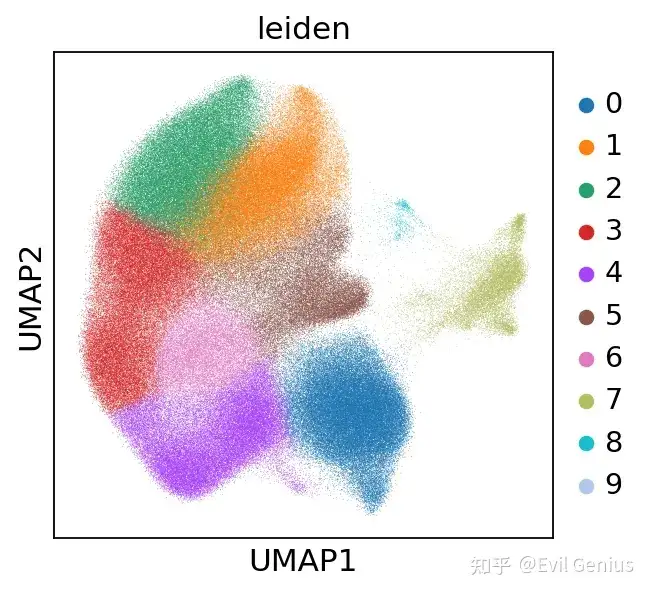
sc.pl.spatial(adata, color='leiden')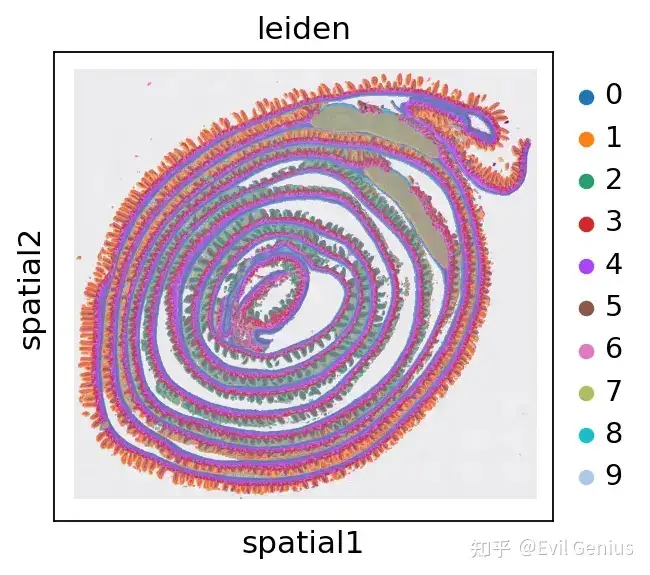
sc.set_figure_params(dpi_save=300, figsize=(12, 9))
sc.settings.figdir = "./results/visium-hd/mouse-intestine/config-8um/"
sc.pl.spatial(adata, color='leiden', frameon=False, save='_leiden_8um.svg')
sc.pl.umap(adata, color='leiden', frameon=False, save='_leiden_8um.svg', show=False)
# sc.pl.spatial(adata, color='leiden',)
sc.pl.spatial(adata, color='leiden', groups=['6', '7'], img_key=None)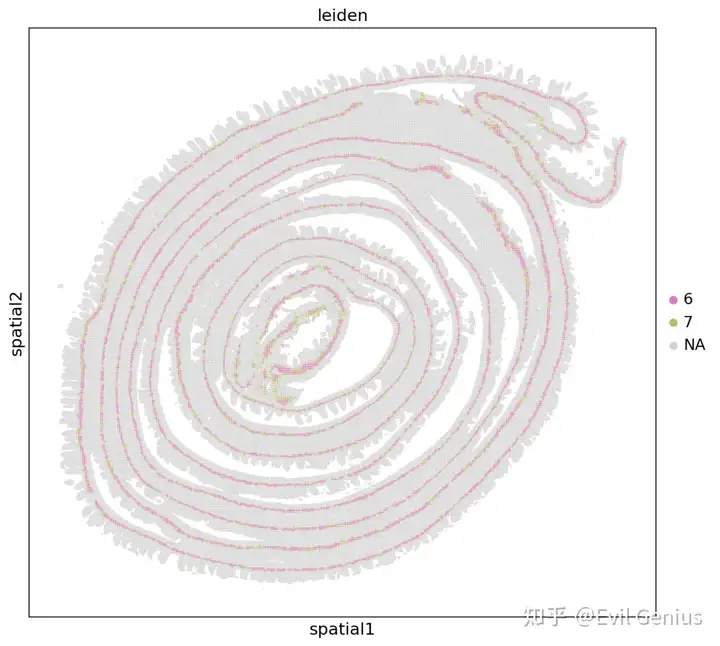
adata.obs['leiden'] = adata.obs['leiden'].astype(str).apply(lambda x: '0' if x in ['10', '11'] else x)
adata.obs['leiden'] = pd.Categorical(adata.obs['leiden'])
sc.pl.umap(adata, color='leiden', show=False, save='_leiden_8um.svg')
sc.pl.spatial(adata, color='leiden', frameon=False, show=False, save='_leiden_8um.svg')
sc.tl.rank_genes_groups(adata, groupby='leiden', method='wilcoxon', dendrogram=False, use_raw=True)
sc.tl.dendrogram(adata, groupby='leiden')
sc.settings.figdir = "./results/visium-hd/mouse-intestine/config-8um/"
sc.pl.rank_genes_groups_matrixplot(adata,
groupby='leiden',
values_to_plot='logfoldchanges',
cmap='RdBu_r',
use_raw=True,
vmin=-4, vmax=4, save="8um_ct.svg")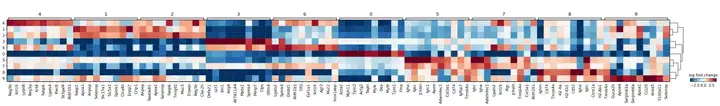
生活很好,有你更好
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录