使用R语言的phytools包在进化树上标记自己测序取样的样本
使用R语言的phytools包在进化树上标记自己测序取样的样本
用户7010445
发布于 2024-04-30 19:38:44
发布于 2024-04-30 19:38:44
要在进化树上标记取样的样本
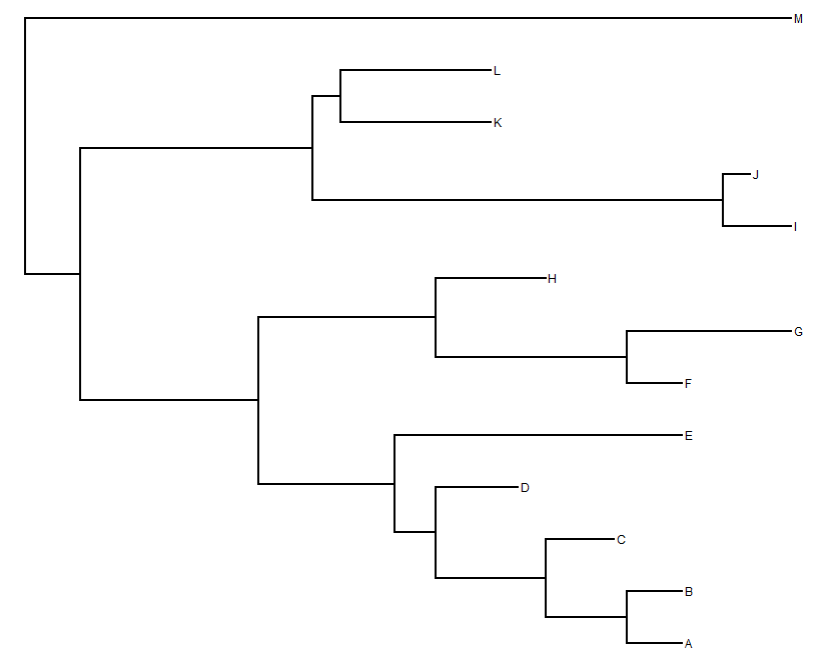
总样本的进化树,这个是随便构造的
library(ggtree)
library(phytools)
tree <- read.tree("data/20220829/fig2.nwk")
plotTree(tree)
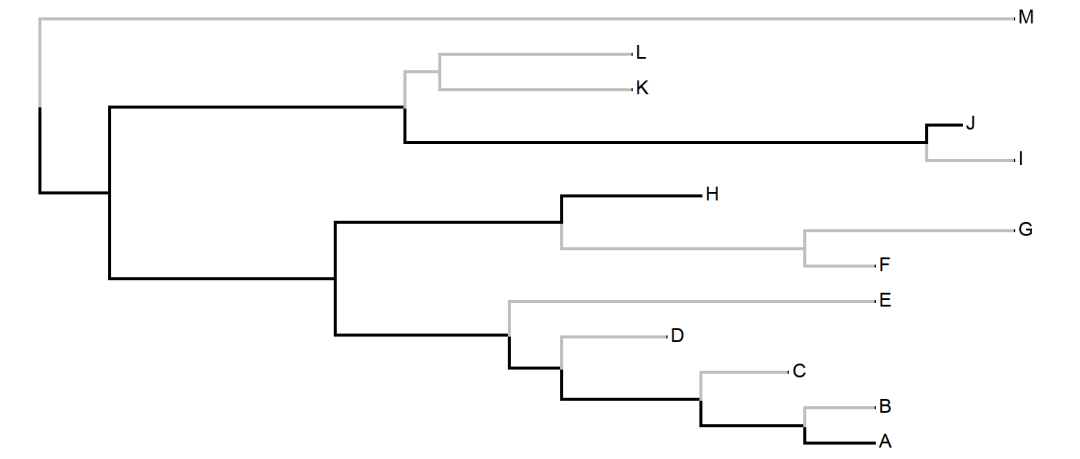
假如我选了 "A","H","J" 这三个样本要测序
获取这这三个样本的 node label
lapply(c("A","H","J"),grep,tree$tip.label) -> tips
获取这三个样本的所有父节点
lapply(tips,phangorn::Ancestors,x=tree,type="all") %>%
unlist() %>% unique() -> parents
c(tips %>% unlist(),parents) -> nodes
## 可以把根节点去掉
setdiff(nodes,Ntip(tree)+1) -> nodes
在进化树上进行标记
paintBranches(tree,
nodes,
state = "1",
anc.state = "0") -> tree03
画进化树
cols<-setNames(c("gray","black"),0:1)
plot(tree03,
lend=1,
add=TRUE,
split.vertical=TRUE,
color=cols)
本文参与?腾讯云自媒体分享计划,分享自微信公众号。
原始发表:2024-04-25,如有侵权请联系?cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读