
AnalyticDB for MySQL是云端托管的PB级高并发低延时数据仓库 通过AnalyticDB for MySQL向量检索功能构建基因检索系统 支持毫秒级针对10亿级别的向量数据进行查询分析 更加快速、高效地为新冠肺炎病毒防控、研发治疗药物以及相关疫苗提供帮助。
1.背景信息2019年年底 中国的新兴商业中心武汉 爆发了一种名为新型冠状病毒肺炎 简称新冠肺炎 的疫情。在新冠肺炎流行的两个多月中 中国造成了3300多人死亡 8万2千多人感染。随着疫情的进一步蔓延 目前已经横跨了109个国家 造成了80多万人感染 4万多人失去了生命。到目前为止 疫情使得50多个国家停摆 全世界范围内造成了数千亿美元的经济损失。在疫情防治期间 阿里云提供了高效基因序列检索技术 助力新冠肺炎病毒序列分析。
2.基因序列检索技术应用范围和现状对于当下疫情 基因序列检索技术主要应用于以下场景
用于新冠肺炎的溯源和分析 帮助疫情防控找到病毒宿主 做好有效防范。基于当下疫情的快速蔓延 当前的基因匹配算法太慢 迫切需要高效匹配算法进行基因序列检索。阿里云AnalyticDB for MySQL团队将基因序列片段转化成对应的1024维特征向量 将两个基因片段的匹配问题 转换成了两个向量间的距离计算问题 从而大大降低了计算开销 实现毫秒级返回相关基因片段 完成基因片段的首次筛选。然后 使用基因相似计算BLAST算法 完成基因相似度的精确排查 从而高效率完成基因序列的匹配计算。匹配算法从原来O(M N)的复杂度降低到O(1)。同时 阿里云AnalyticDB for MySQL提供强大的机器学习分析工具 通过基因转向量技术 将局部的和疾病相关的关键靶点基因片段转成特征向量 用于基因药物的研发 大大加速了基因分析过程。
3. AnalyticDB for MySQL基因检索系统新冠肺炎病毒的RNA序列可以用一串核酸序列 又称碱基序列 表示 RNA序列含有四种核苷酸 分别用A、C、G和T表示 分别代表腺嘌呤、胞嘧啶、鸟嘌呤、胸腺嘧啶。每个字母代表一种碱基 无间隔排列在一起。每一个物种的RNA序列均不相同但又有规律 基因检索系统可以通过输入一串病毒的基因片段 检索相似的基因 用来对病毒的RNA序列进行分析。
为方便演示AnalyticDB for MySQL基因片段检索方法 我们从GenBank下载了大量病毒的RNA片段 并将GenBank内部关于病毒的论文以及Google Scholar中相关病毒的论文导入AnalyticDB for MySQL基因检索数据库中。
AnalyticDB for MySQL基因检索的演示界面如下图所示 将新冠肺炎病毒的序列 COVID-19 上传到AnalyticDB for MySQL基因检索系统中 AnalyticDB for MySQL基因检索系统只需几毫秒即可检索到相似的基因片段 当前示例系统只返回匹配度超过0.8的基因片段 。从返回的基因片段得出穿山甲携带的新冠肺炎病毒(GD/P1L)、蝙蝠携带的新冠肺炎病毒(RaTG13)以及SARS和MARS病毒 其中GD/P1L的序列匹配度最高为0.974 由此推断出新冠肺炎病毒很可能是通过穿山甲传染到人的。
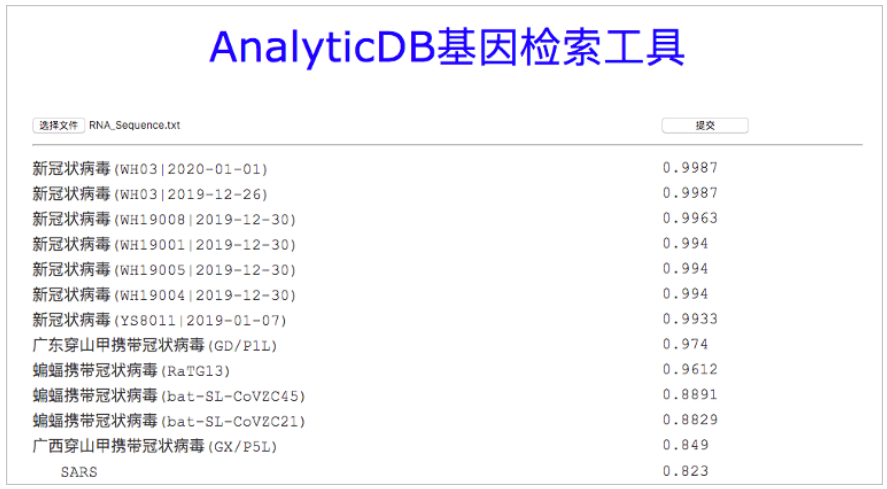
图1 AnalyticDB for MySQL基因检索演示界面
如果RNA片段非常相似 说明这两个RNA可能有相似的蛋白质表达和结构。通过基因检索工具 可以看到SARS和MARS与新冠肺炎病毒的匹配度为0.8以上 说明可以将一些SARS或者MARS的研究成果应用到新冠肺炎病毒上。系统提取了每种病毒的论文 通过文本分类算法 将论文划分为检测类、疫苗类和药物类 例如下图为SARS的七种检测方法、四种疫苗接种方法和10中治疗药物。其中 对SARS有效的荧光定量PCR检测 目前正应用于新冠肺炎病毒的检测 基因疫苗的方法以及诱导体内免疫疫苗的方法 也正在展开研究 治疗药物中瑞德西韦以及相关的干扰素也都用于新冠肺炎病毒的治疗上。

图2 SARS病毒相关论文检索分类结果
单击上图干扰素链接可以查看相关论文 当前系统调用了自动翻译软件 抽取中文版文件名关键词作为文件名 方便用户阅读。
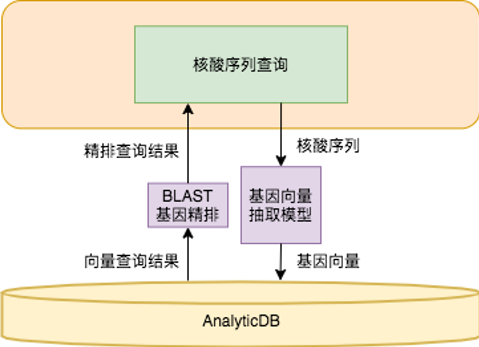
图4 基因检索实现架构
AnalyticDB for MySQL基因检索系统中 AnalyticDB for MySQL负责存储和查询所有结构化数据 例如基因序列的长度 长度包含基因的论文名称、基因种类、DNA或者RNA等 和基因序列产生的特征向量。查询数据时 通过基因向量抽取模型将基因转化成向量 在AnalyticDB for MySQL向量数据库中进行粗排检索 然后在返回的向量匹配结果集中使用经典的BLAST算法进行精确检索 返回最相似的基因序列。
AnalyticDB for MySQL基因检索系统的核心是基因向量抽取模型 该模块可以将核苷酸序列转化成向量。目前AnalyticDB for MySQL抽取了各种病毒的RNA全部序列样本进行训练 可以非常方便地对病毒的RNA进行相似度计算。同时 基因向量抽取模型也可以扩展应用于其他物种基因检索。
5.基因向量抽取算法目前词向量技术已经非常成熟 被广泛应用于机器翻译、阅读理解、语义分析等相关领域 并取得了巨大成功。词向量化采用了分布式语义的方法表示一个词的含义 一个词的含义就是这个词所处的上下文语境。例如高中英语中的完形填空题 一篇短文空出10个地方 学生根据空缺词的上下文语境选择合适的词。也就是说上下文语境能够准确地表达这个词 如果某位同学选择了正确的选词 表示该同学理解了空缺词的含义。因此 通过上下文词的关系 采用词向量算法 可以为每个词生成一个向量 通过计算两个词向量之间的相似度 得到两个词的相似度。
同样的道理 基因序列的排列具有一定的规律 并且每一部分基因序列所表达的功能和含义不同。可以将很长的基因序列划分成小的单元片段 也就是词 进行分析 并且这些词也有上下文语境 这些词相互连接、相互作用共同完成相对应的功能 形成合理的表达。因此 生物科学家们采用词向量算法对基因序列单元进行向量化 两个基因单元相似度很高 说明需要这两个基因单元共同来表达和完成相应的功能。
总体而言 AnalyticDB for MySQL基因向量抽取算法分为三步
1 在氨基酸序列中定义词。生物信息学中用K-mers来分析氨基酸序列 K-mer是指将核酸序列分成包含K个碱基的字符串 即从一段连续的核酸序列中迭代选取长度为K个碱基的序列 若核酸序列长度为L K-mer长度为K 那么可以得到L-K 1个K-mers。例如下图所示 假设某序列长度为12 设定选取的k-mer长度为8 则得到 12-8 1 5 5个5-mers。这些k-mer 就是氨基酸序列中的词。
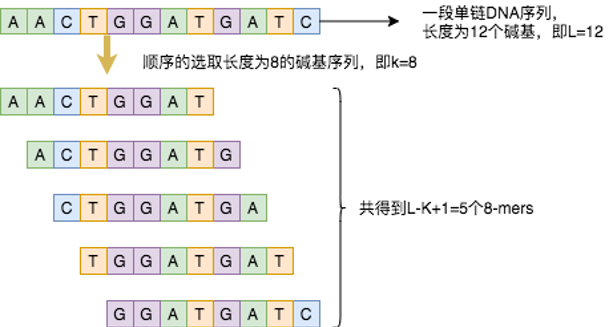
图5 8-mers核酸序列示意图
2 找到氨基酸序列的上下文语境 将基因序列中的词转换成1024维向量。对于词向量算法而言 另一个重要的问题就是上下文的语境。AnalyticDB for MySQL基因向量抽取算法在氨基酸片段中选择一个长度为L的窗口 该窗口内的氨基酸片段可认定为在同一语境内。例如CTGGATGA是一段核酸序列 选取了长度为10的窗口 AnalyticDB for MySQL基因向量抽取算法将CTGGATGA转换成5个5-mers即{AACTG, ACTGG, CTGGA, GGATG, GATGA}。对于其中一个5-mer{CTGGA}而言 另外四个{AACTG, ACTGG, GGATG, GATGA}5-mers就是当前5-mer {CTGGA}的上下文语境。
AnalyticDB for MySQL基因向量抽取算法套用词向量空间训练模型 对已有生物基因的K-mers进行训练 便可将一个K-mer 基因序列中的一个词 转换成1024维向量。
3 类似于词向量模型 K-mer向量模型也拥有和词向量模型相似的数理计算性质。公式一说明核苷酸序列ACGAT的向量减去GAT序列的向量和AC序列的向量距离非常接近。公式二说明核苷酸序列AC的向量加上ATC序列的向量和ACATC序列的向量距离也很接近。因此 根据这些数理特征 计算一个长氨基酸序列向量时 可以将这个序列中每一段的K-mer序列进行累加 最后进行归一化就能得到整个氨基酸序列的向量。同时 为提升精度 可以将基因片段看作一个文本 使用doc2vec4将整个序列转换成向量进行计算。为进一步验证算法性能 AnalyticDB for MySQL基因向量抽取算法计算了常用于基因检索库中的BLAST算法序列与基因转向量l2距离序列的相似度 两个序列的斯皮尔曼等级相关系数是0.839。以上得出结论 将DNA序列转换成向量用于相似基因片段的初次筛选 是有效且可行的。
6.向量检索功能概述一般包含向量检索的应用系统中 开发者通常会使用向量检索引擎 例如Faiss 存储向量数据 然后使用关系型数据库存储结构化数据。因此 查询时也需要交替查询两个系统 明显额外增加了开发人员的工作量 数据查询性能也不是最优。
AnalyticDB for MySQL是云端托管的PB级高并发低延时数据仓库 可以毫秒级针对10亿级别的向量数据进行查询 100毫秒级别的响应时间。AnalyticDB for MySQL全面兼容MySQL协议以及SQL:2003语法标准 其向量检索功能支持对图像、文本推荐、声纹、核苷酸序列等相似性进行查询和分析 目前在多个城市的安防项目中已大规模部署了AnalyticDB for MySQL。
AnalyticDB for MySQL支持结构化和非结构化数据的近似检索和分析 通过SQL接口即可快速搭建基因检索或者基因 结构化数据混合检索等系统。在混合检索场景中AnalyticDB for MySQL的优化器会根据数据的分布和查询条件选择最优执行计划 在保证数据召回率的同时 得到最优的性能。例如 通过以下一条SQL即可检索RNA核酸序列。
-- 查找RNA和提交的序列向量相近的基因序列。 select title, # 文章名 length, # 基因长度 type, # mRNA或DNA等 l2_distance(feature, array[-0.017,-0.032,...]::real[]) as distance # 向量距离 from demo.paper a, demo.dna_feature b where a.id b.id order by distance; # 用向量相似度排序
上述SQL中表demo.paper用于存储上传的每篇文章的基本信息 demo.dna_feature存储各个物种的基因序列对应的向量。通过基因转向量模型 将要检索的基因转成向量[-0.017,-0.032,...] 然后在AnalyticDB for MySQL数据库中进行检索。
当前系统也支持结构化信息 非结构化信息 核苷酸序列 的混合检索 例如查找和冠状病毒相关的类似基因片段时 只需要在SQL中增加where title like %COVID-19% 即可。
本文参考的相关文献 感兴趣的读者可访问这里或扫描下方二维码了解详情。

如今,几乎每个基础设施供应商都将混合云或多云作为一种产品采用。采用多个云服...
在进行数据分析与可视化之前,得先处理好数据,而很多时候需要处理的都是文本数...
目前,市面上有许多代码分析工具,但昂贵的费用对于初创公司和个人来说有些难以...
来源 | 阿里巴巴云原生公众号 3 月 20 日,Nacos 2.0 正式发布。Nacos 是阿里巴...
本文已经过 dmitripavlutin 授权翻译! 除了普通对象之外,数组是 JavaScript 中...
想人在家中就能切身一览这世界各个地方的景色?想点点屏幕就能如同亲临般逛街购...
显然,数据现在是我们所有生活的核心,在过去十年中,智能手机的蓬勃 发展推动了...
在上一章节中已经对数据结构的基本概念有了了解,主要就是数据结构研究的三个方...
3月5日 记者采访获悉 江门农商银行日前完成自助分析平台的升级换代 由阿里云云原...
#今年双十一快递有多快#、#双十一快递比外卖还快# 这些话题在今年双十一期间频繁...