化脓性汗腺炎和银屑病单细胞数据复现2-小提琴图复现
上次推文更新了一篇文章复现,今天同样是接着之前那篇文章来做一下小提琴图的复现。 上次推文链接:日光性皮炎和银屑病单细胞数据集复现 同时来一个纠错,之前写的日光性皮炎应该是化脓性汗腺炎和 HIS 修改为 HS


library(ggsci)
library(dplyr)
library(Seurat)
library(cowplot)
library(data.table)
library(dplyr)
library(ggplot2)
library(stringr)
getwd()
sce.all=readRDS("./2-harmony/sce.all_int.rds")
sce.all.int=sce.all
celltype=data.frame(ClusterID=0:18 ,
celltype= 0:18)
#定义细胞亚群
celltype[celltype$ClusterID %in% c( 2,4,5,7,8,11,12,15,16 ),2]='Keratinocyte'
celltype[celltype$ClusterID %in% c( 13 ),2]='Fibroblast'
celltype[celltype$ClusterID %in% c( 9 ),2]='DC-2'
celltype[celltype$ClusterID %in% c( 3 ),2]='DC-1'
celltype[celltype$ClusterID %in% c( 0,1),2]='T'
celltype[celltype$ClusterID %in% c( 10),2]='melanocytes'
celltype[celltype$ClusterID %in% c( 17),2]='cycling'
celltype[celltype$ClusterID %in% c( 14),2]='B'
celltype[celltype$ClusterID %in% c( 6 ),2]='plasma'
celltype[celltype$ClusterID %in% c( 18),2]='Mast'
head(celltype)
celltype
table(celltype$celltype)
sce.all.int@meta.data$celltype = "NA"
for(i in 1:nrow(celltype)){
sce.all.int@meta.data[which(sce.all.int@meta.data$RNA_snn_res.0.8 == celltype$ClusterID[i]),'celltype'] <- celltype$celltype[i]}
Idents(sce.all.int)=sce.all.int$celltype
table( Idents(sce.all.int))
sel.clust = "celltype"
sce.all.int <- SetIdent(sce.all.int, value = sel.clust)
table(sce.all.int@active.ident)
p_umap=DimPlot(sce.all.int, reduction = "umap", group.by = "celltype",label = T,label.box = T)
p_umap
p2=VlnPlot(sce.all.int, group.by = "celltype", split.by = "group",features = "IL1B", pt.size = 0, ncol = 3, same.y.lims=T) +
scale_y_continuous(breaks=seq(0, 100, 5)) +
NoLegend()
p2
#修正上次推文的HIS为HS
sce.all.int$group<-ifelse(grepl("GSM6840117|118|119|120|121|122|123|124",sce.all$orig.ident),"HS" ifelse(grepl("GSM6840143|144|145|146|147|148|149|150|151|152",sce.all$orig.ident),"Control", "Psoriasis"))
table(sce.all.int$group)
#因子改变分组顺序
sce.all.int$group<-factor(sce.all.int$group,levels=c("Control","Psoriasis","HS"))
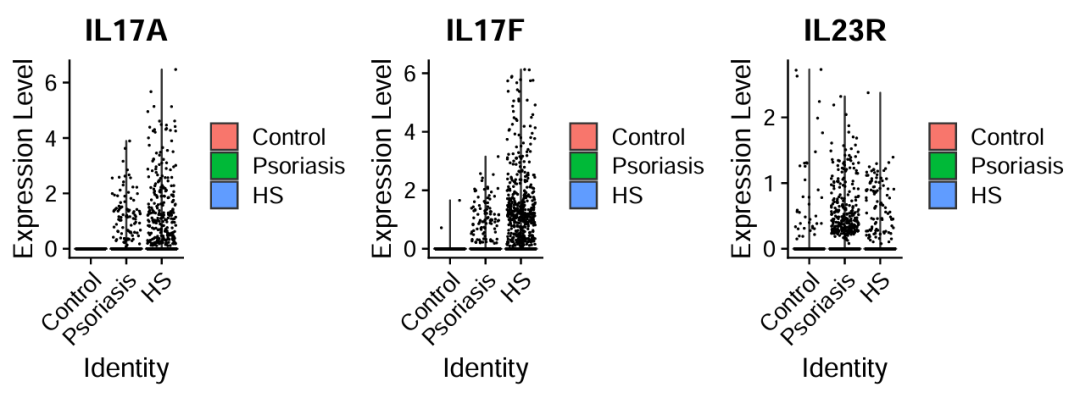
pp1<-VlnPlot(sce.all.int,group.by = "group", "IL17A")+ theme(axis.text.x=element_text(angle=45,hjust = 1));pp1
pp2<-VlnPlot(sce.all.int,group.by = "group", "IL17F")+ theme(axis.text.x=element_text(angle=45,hjust = 1));pp2
pp3<-VlnPlot(sce.all.int,group.by = "group", "IL23R")+ theme(axis.text.x=element_text(angle=45,hjust = 1));pp3
library(patchwork)
pp1+pp2+pp3+plot_layout(ncol = 3)
ggsave(file="vplot_1.pdf",width = 8,height = 3)
pp<-VlnPlot(sce.all.int,group.by = "group", "IL1B")+ theme(axis.text.x=element_text(angle=45,hjust = 1));pp
pp4<-VlnPlot(sce.all.int,group.by = "celltype", split.by = "group", "IL1B")+
theme(axis.text.x=element_text(angle=45,hjust = 1));pp4
ggsave(file="vplot_2.pdf",width = 10,height = 3)
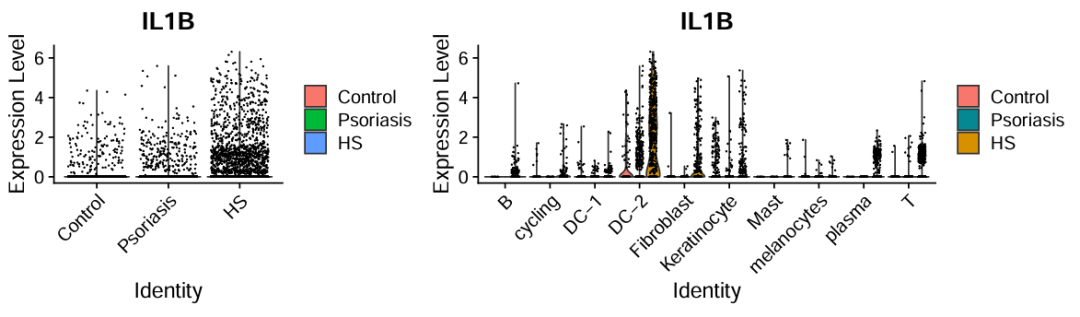
从小提琴图来看整体复现程度还是挺高的。
本文参与?腾讯云自媒体分享计划,分享自微信公众号。
原始发表:2024-04-18,如有侵权请联系?cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读